Visual Programming으로 AI 모델링이 가능한 웹 어플리케이션 개발 후기
- -
Visual Programming으로 AI 모델링이 가능한 웹 어플리케이션을 개발하며

지난 7~8월 여름방학에 고려대학교 정보대학 HVCL 연구소에서 학부 인턴으로 근무하면서 어떠한 일을 진행했는지를 개발 후기를 남기는 게 좋을 것 같아서 글을 쓰려고 한다.
그동안 팀 프로젝트 단위로 개발을 해 본 경험이 있지만, 대학원 연구실에서 근무하는 것은 처음이었고 게다가 개인적으로 의학 분야는 처음 접해서 많이 애를 먹었다.
하지만 막상 두 달의 근무 기간이 끝나고 회고해보면 적지 않은 성장을 이룬 것 같아서 보람찬 마음이 크고, 앞으로 Backend와 AI Engineering이라는 나의 궁극적인 목표에 한 발자국 다가갈 수 있는 초석을 다졌다고 생각한다.
두 달이라는 짧은 시간동안 AI 모델링과 웹 어플리케이션 개발에 익숙하지 않은 인턴 둘이 프로젝트를 진행했기에 아직은 프로토타입에 그치지만, 그래도 기초적인 자료구조를 정의하여 구현하고 전반적인 뼈대가 되는 베이스라인을 설계했다는 점에서 개인적으로 가졌던 기대보다 더 나은 결과가 나왔다고 말할 수 있을 것 같다.
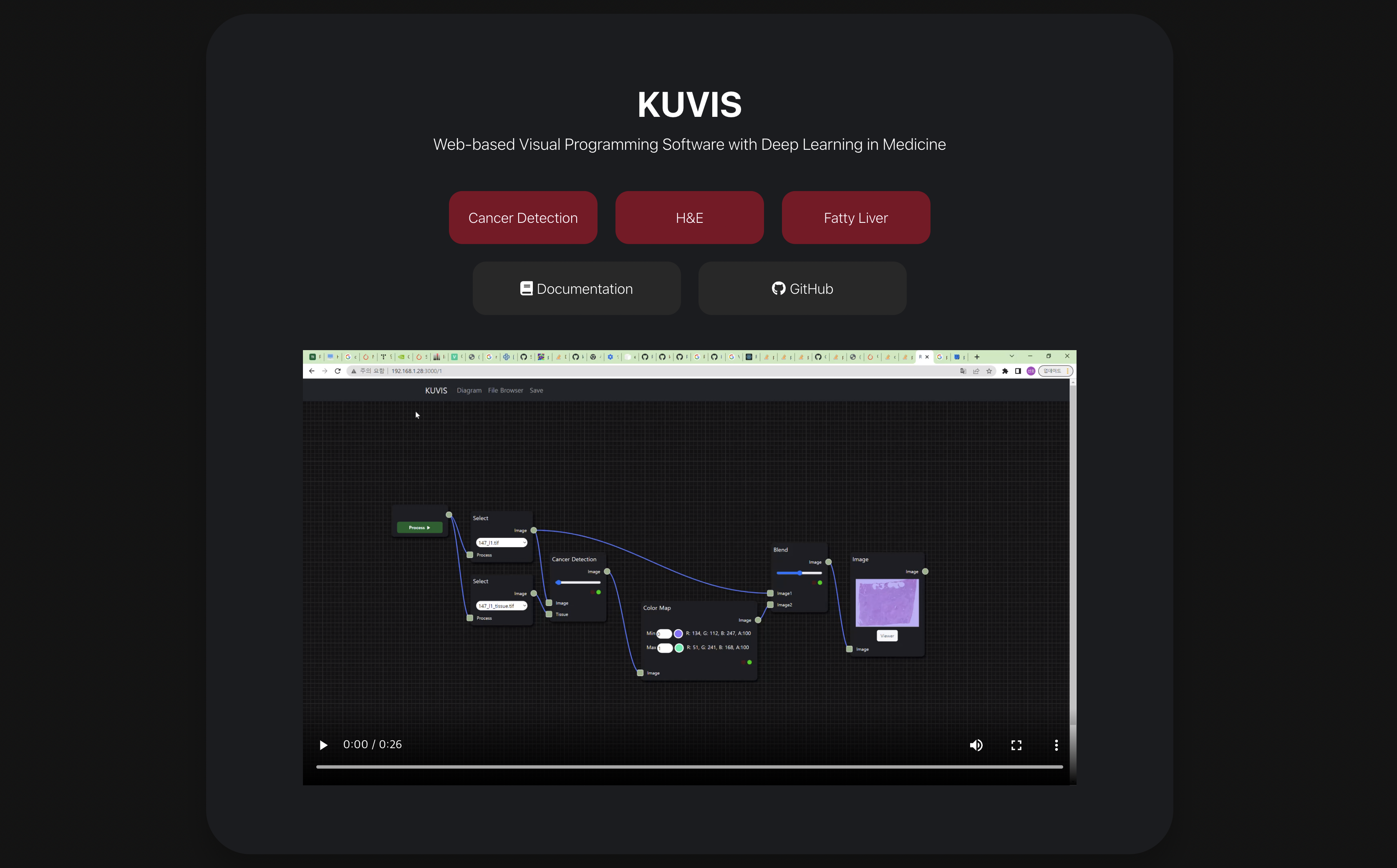
같이 프로젝트를 담당했던 mintway0341님의 프론트엔드 실력이 뒷받침되지 않았다면 지금처럼 퀄리티가 보장된 웹 어플리케이션이 프로토타입으로 나오지 못했을 텐데, 내 백엔드 실력이 많이 부족함에도 불구하고 프론트 엔드를 끝까지 맡아준 mintway0341님께 감사한 마음을 전하고 싶다.
그리고 프로젝트를 전반적으로 매니징하고 방향을 제시해주신 교수님과 사수분들께도 이 글을 빌려 감사하다는 말을 드리고 싶다.
프로그램의 특징
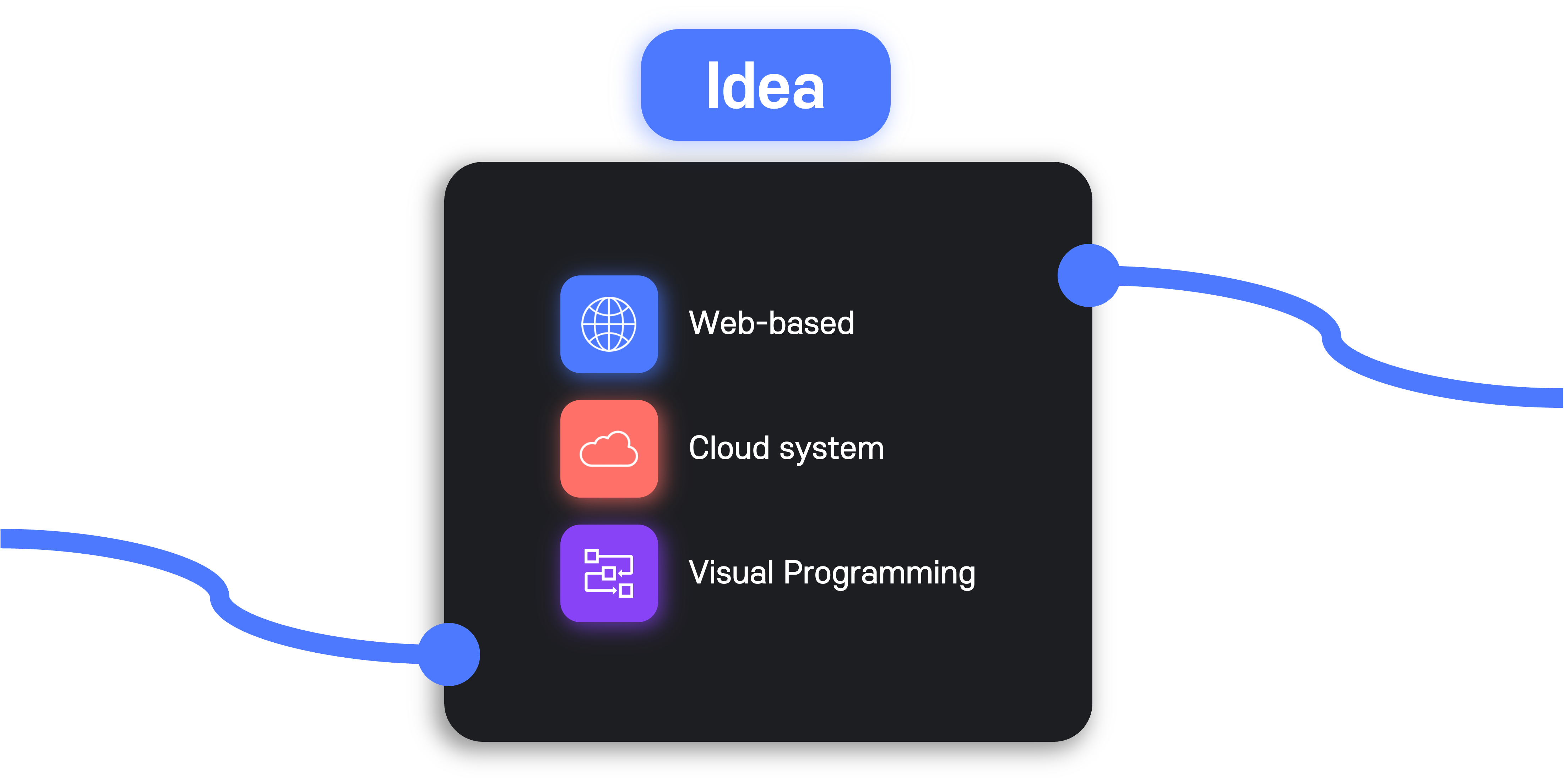
이번 프로젝트의 전반적인 특징을 세 가지로 요약하면 다음과 같다.
- 인터넷 접속이 가능한 환경이면 언제든지 사용할 수 있는 웹 어플리케이션이다.
- 사용자가 작업한 결과물을 어디에서든지 확인하고 수정 가능한 클라우드 시스템 기반이다.
- 코드 작성이 아닌 시각적으로 workflow를 구성할 수 있는 비주얼 프로그래밍을 지원한다.
클라우드 시스템을 기반으로 하는 웹 어플리케이션도 적지 않은 비중을 차지하는 장점이지만, 이중에서 먼저 세 번째 특징부터 설명해보고자 한다.
비주얼 프로그래밍(Visual Programming) 플랫폼을 만들다
이번에 참여한 프로젝트는 의학 영상 자료 분석과 AI 모델링을 비주얼 프로그래밍(Visual Programming)으로 수행할 수 있는 웹 어플리케이션을 개발하는 것이 목표였다.
비주얼 프로그래밍이란 잠시 위키피디아의 '비주얼 프로그래밍 언어(Visual Programming Language)'의 정의를 인용하자면 다음과 같다.
사용자가 텍스트로 직접 작성하는 대신에 그래피적으로 프로그램 요소를 조작하여 프로그램을 개발할 수 있게 하는 모든 종류의 프로그래밍 언어를 일컫는다.
이번에 개발한 프로젝트도 위의 정의에 입각하여 의학 영상 자료를 분석하고 연구하지만 소프트웨어 분야에 비전공자인 사람도 자신이 구상하는 DAG(Directed Acyclic Graph) workflow를 diagram으로 손쉽게 설계하여 바로 실행할 수 있는 비주얼 프로그래밍의 일종이다.
또한 비전공자 뿐만이 아니라 전공자도 자신이 원하는 workflow를 코드로 관리하는 것보다 시각적으로 한눈에 확인할 수 있으며, 여러 사람이 동시에 AI 모델링을 수행하거나 영상 자료를 분석할 때 다른 사람이 작성한 코드를 가져와서 읽는 것보다는 workflow diagram을 확인하는 것이 업무의 효율성을 높일 수 있는 방법이라고 생각했다.
비주얼 프로그래밍 작업 환경을 제작하기 위해 가장 크게 고민했던 부분은 어떤 라이브러리를 사용하는지였다.
사실 두 달 안에 프론트엔드의 모든 것까지 직접 다 일일이 구현하는 건 거의 불가능에 가까웠고, 기존에 존재하는 라이브러리를 활용하면서 정해진 제약 시간 안에 최대한 퀄리티 높게 제작할 수 있는 방법을 찾아야 했다.
여러 라이브러리와 프레임워크를 탐색했고, 그중에서 Rete.js 프레임워크가 가장 좋은 것으로 판단되어 이를 비주얼 프로그래밍을 위한 workspace 환경을 구현하는 용도로 사용했다.
Rete.js
rete.js.org
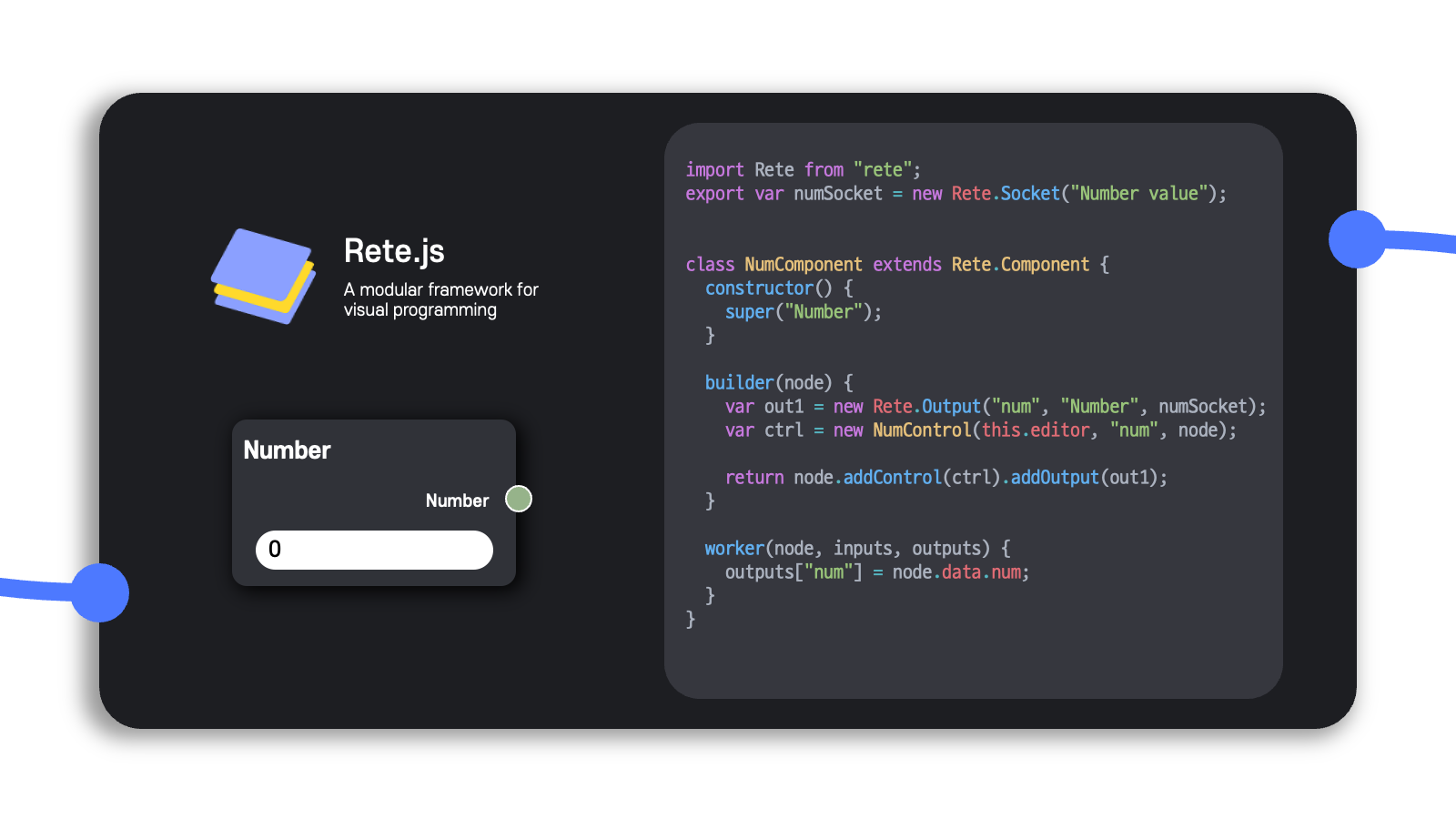
Rete.js의 장점은 기본적으로 내재되거나 이미 구현된 기능들이 많고, JavaScript에 익숙한 사람이라면 코드를 다룰 때 문법적으로 친근하고 다루기 용이하다는 것이다.
그러나 단점은 기존의 React를 사용할 때처럼 함수 형식으로 작성하기보다는 대부분이 클래스를 정의하여 사용하는 코드가 많아서 기존의 코드 작성 스타일과는 다르고, 좀 더 깊숙히 파고들어서 분석하려면 클래스의 상속관계를 면밀히 파악해야 하는 점이었다.
사용하기에 직관적이고 간단하다
사용자가 사용 방법을 직관적으로 익히고 사용할 수 있도록 만드는 데 노력을 기울였으며, 실제로 이번 프로젝트의 프로토타입을 직접 다뤄보면 사용법이 간단하다는 점을 느낄 수 있다.
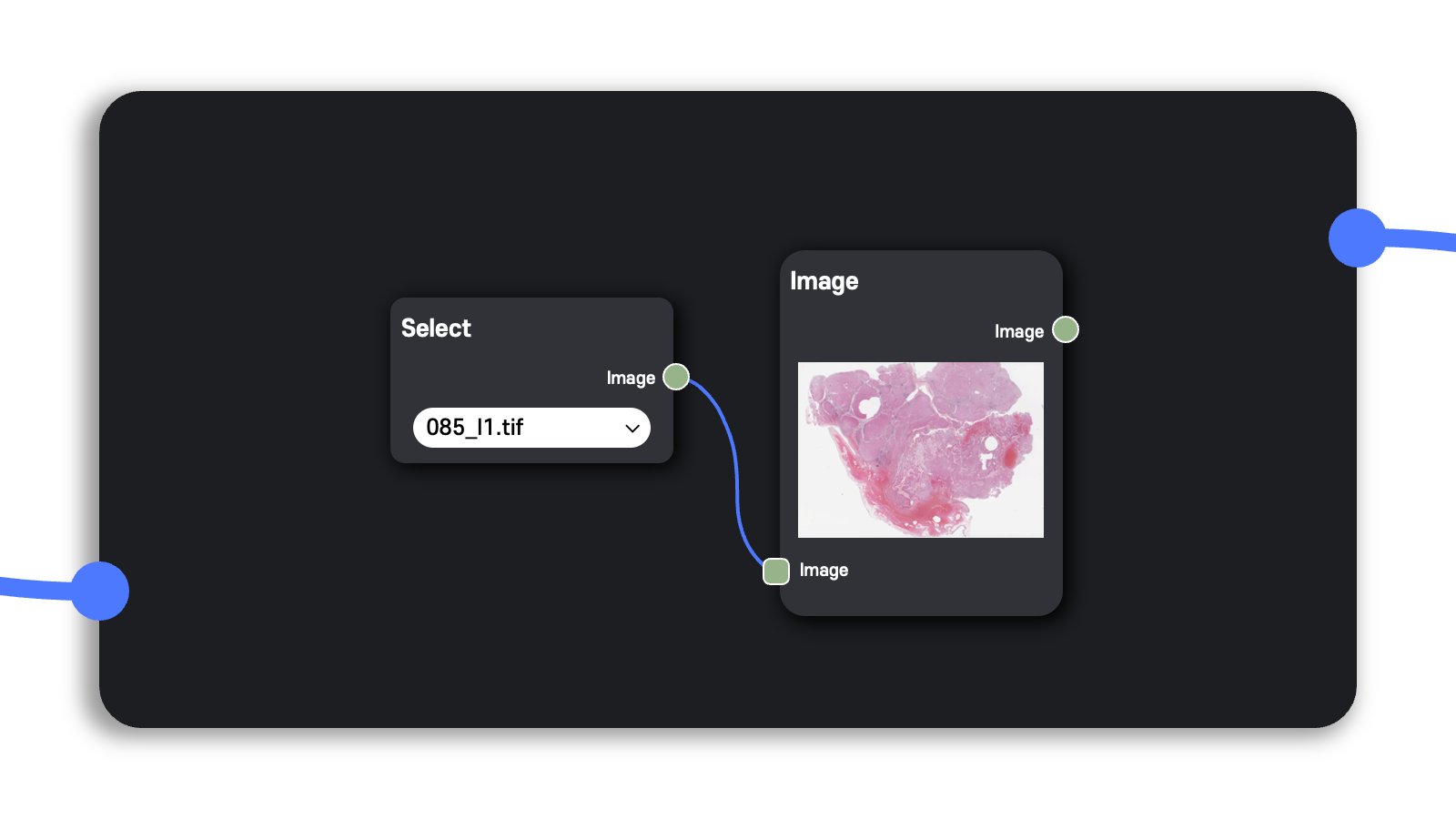
처음 어플리케이션에 브라우저로 접속했을 때 격자 무늬의 회색 빈 화면인 workspace가 뜨는데, 여기에 사용자가 원하는 node를 추가하고, node 간 edge를 연결하여 실행하고 싶은 DAG(Directed Acyclic Graph) workflow를 구성할 수 있다.

Node를 추가할 때는 빈 화면 어느 곳이든 마우스 오른쪽 클릭하여 사용자가 원하는 node를 workspace에 추가할 수 있다.
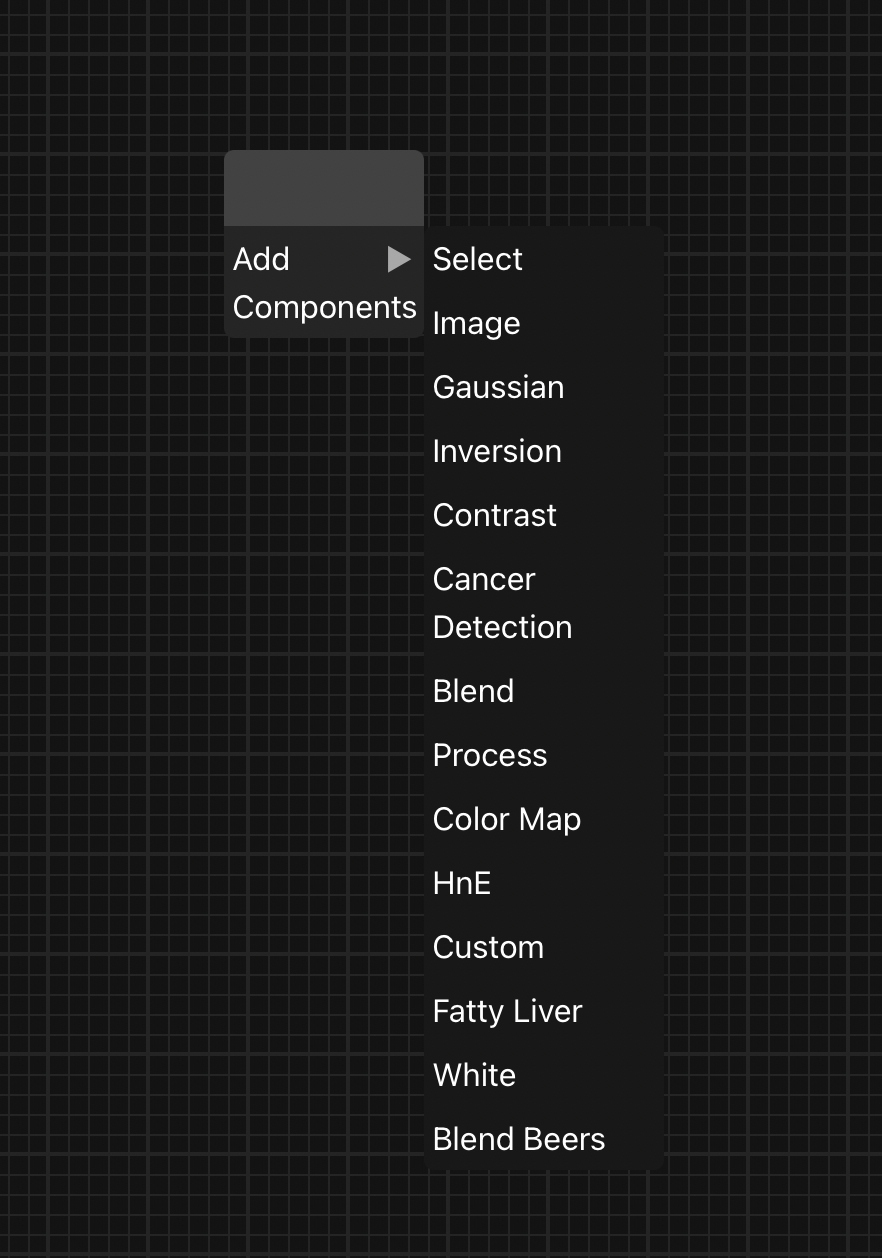
이번 개발 기간동안에는 시나리오를 진행할 14개의 node를 제작했지만, 앞으로 용도에 맞게 종류별로 구분하여 다양한 node를 개발하면 기능을 확장할 수 있을 것으로 기대된다.
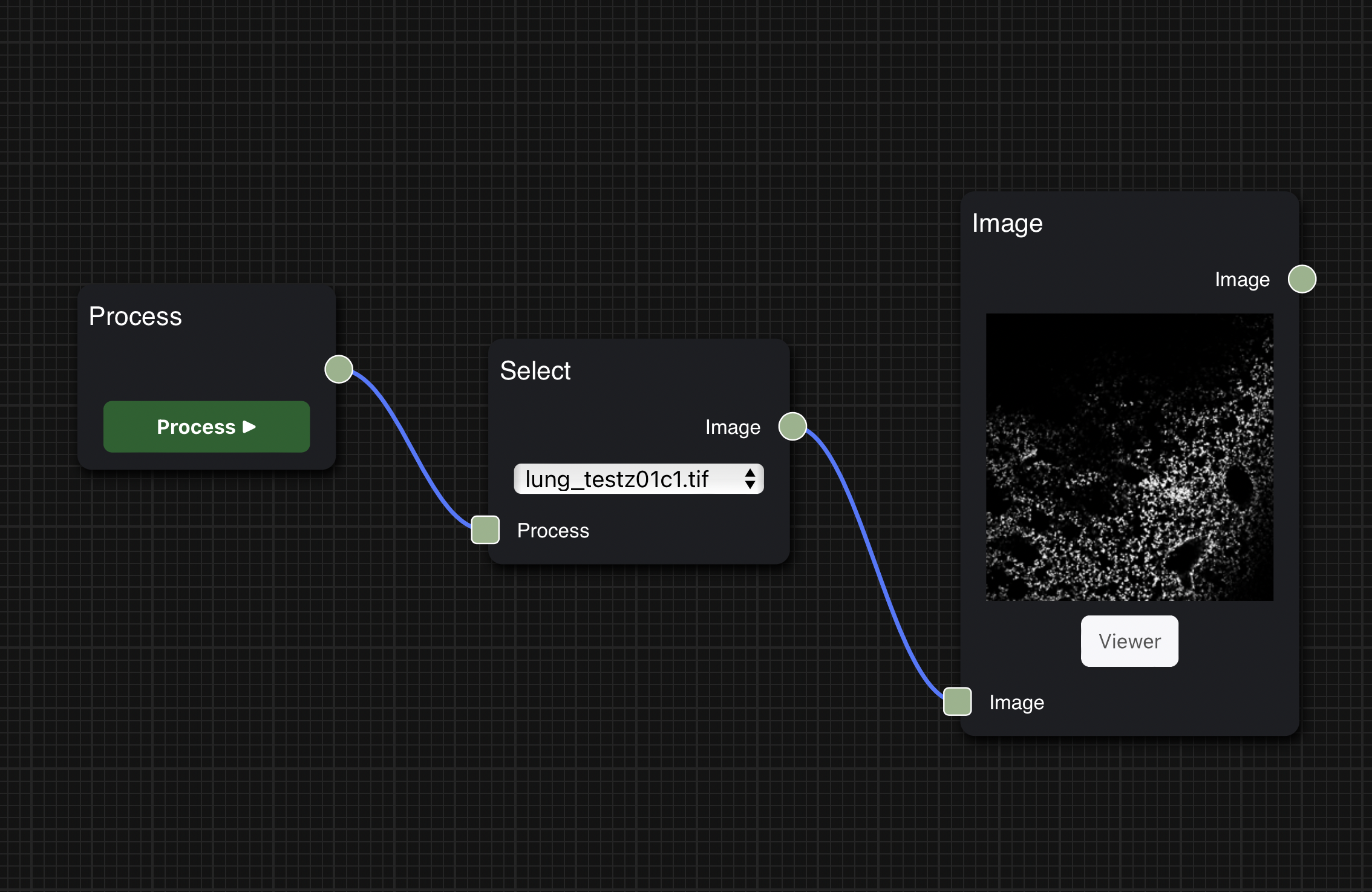
일반적으로 한 Node에서 왼쪽 변의 아래에 위치한 사각형은 input, 오른쪽 변의 중간에 위치한 사각형은 output이다.
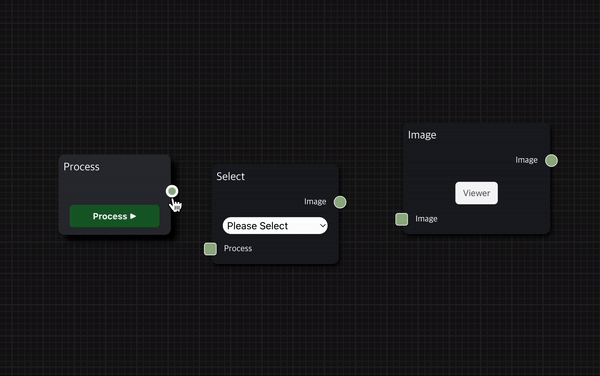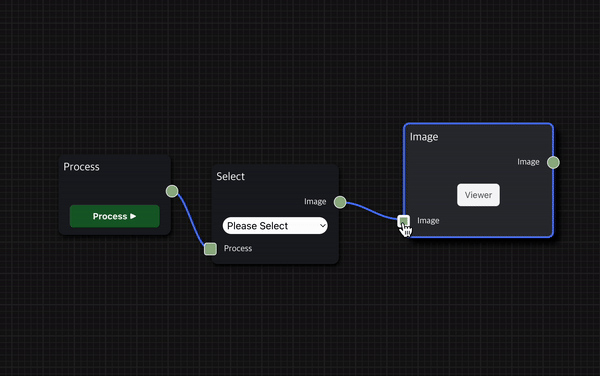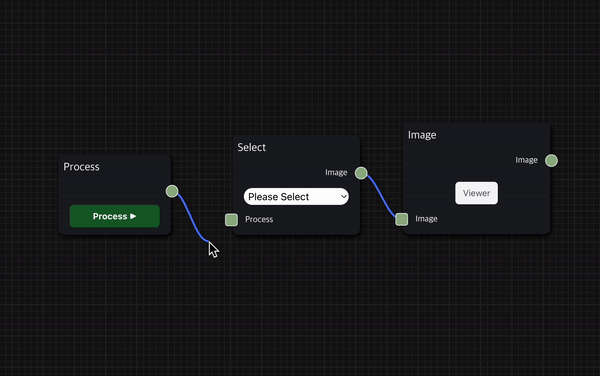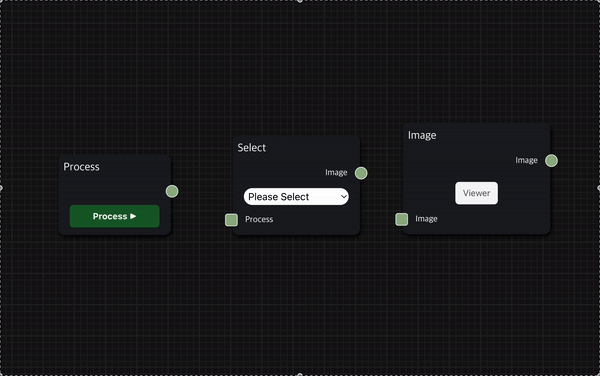
Node끼리 연결하여 workflow를 구성하는 방법도 간단하다.
현재 Node의 input을 이전 Node의 output과 연결하고, 마찬가지로 현재 Node의 output을 다음 Node의 output과 연결하여 workflow를 이어갈 수 있다.
Node의 input 또는 output 영역을 클릭하면 파란색 실선이 생성되어 실선의 끝이 마우스의 위치를 따라가며, 그 상태에서 원하는 node의 output 또는 input 영역을 클릭하여 연결할 수 있다.
그리고 node의 위치를 변경할 수도 있는데, node를 마우스로 드래그하여 workspace에서 원하는 위치에 배치할 수 있다.
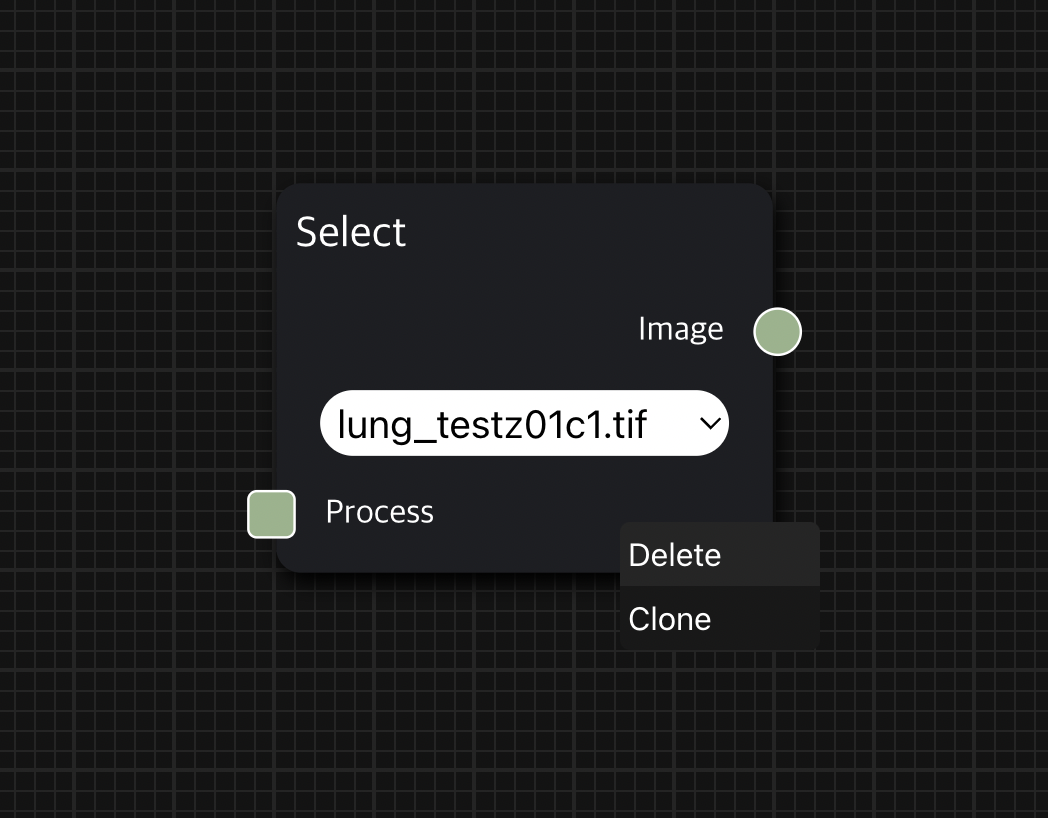
Workspace에 이미 생성한 node를 삭제하는 방법도 간단한데, 삭제하고자 하는 node 위에서 마우스 오른쪽 클릭하면 diagram에서 해당 node를 지울 수 있는 Delete를 수행할 수 있다.
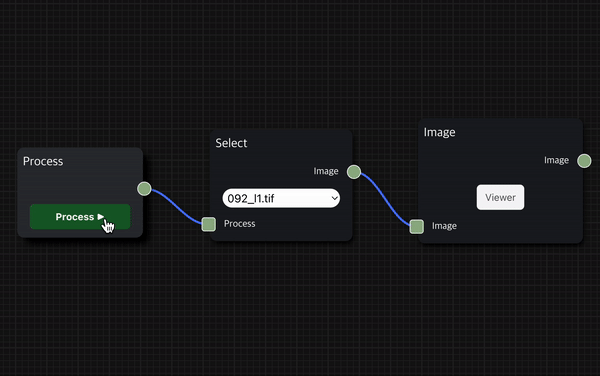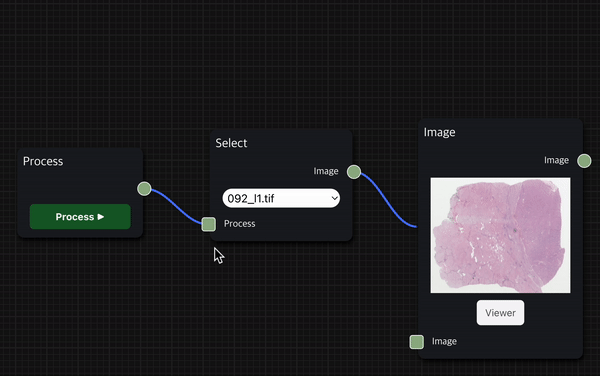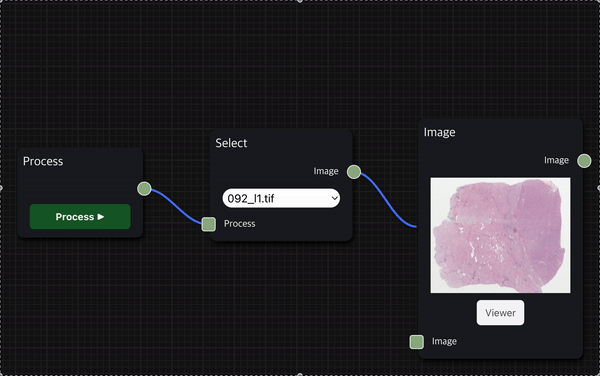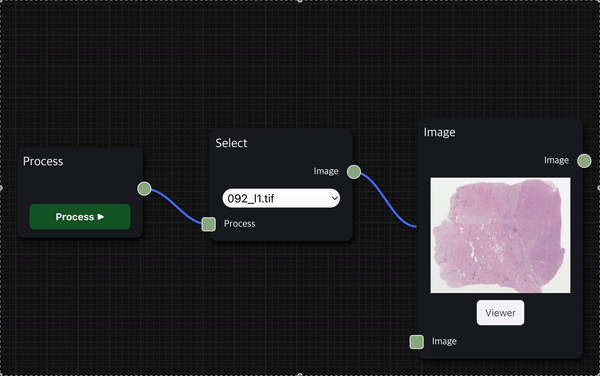
Process Node안의 Process ▶︎ 버튼을 클릭하여 사용자가 실행하기를 원하는 workflow를 실행할 수 있는데, 이를 사용자가 실행하고자 하는 workflow 동작 순서상 가장 앞에 있는 Node와 연결한다.
그리고 Process ▶︎ 버튼을 클릭하면 사용자가 구성한 workflow를 바로 실행할 수 있다.
이처럼 사용법 자체는 쉽고 단순하지만, 이를 바탕으로 앞으로 소개할 다양한 의학 영상 분석 task를 수행할 수 있는 있어서 잠재력이 크다고 생각한다.
인터넷만 접속 가능하면 바로 사용 가능하다
사용자의 컴퓨터에 별도의 응용프로그램 설치를 필요로 하지 않고 인터넷만 있으면 바로 웹 브라우저로 AI 모델링 작업을 할 수 있다는 점도 주목할 만하다.
클라우드 시스템을 기반으로 하므로 고성능을 요하는 작업을 수행한다고 하더라도 사용자의 로컬 컴퓨터의 스펙을 고려할 필요 없이 웹 브라우저만 있으면 된다.
그런데 이를 위해서는 바로 사용자가 이용하는 클라우드 저장소와 별도로 이를 서버에서 가져와서 처리할 수 있는 별도의 저장 공간이 필요해 보였다.
아래에서도 설명하겠지만, 사용자가 클라우드에 저장하는 데이터 자체가 고용량이므로 서버 자체의 메모리에 한꺼번에 올리기보다는 자료구조를 따로 정의하여 이에 맞게 데이터를 처리하고 임시로 저장하는 서버의 별도 저장소가 필요하다고 느꼈다.
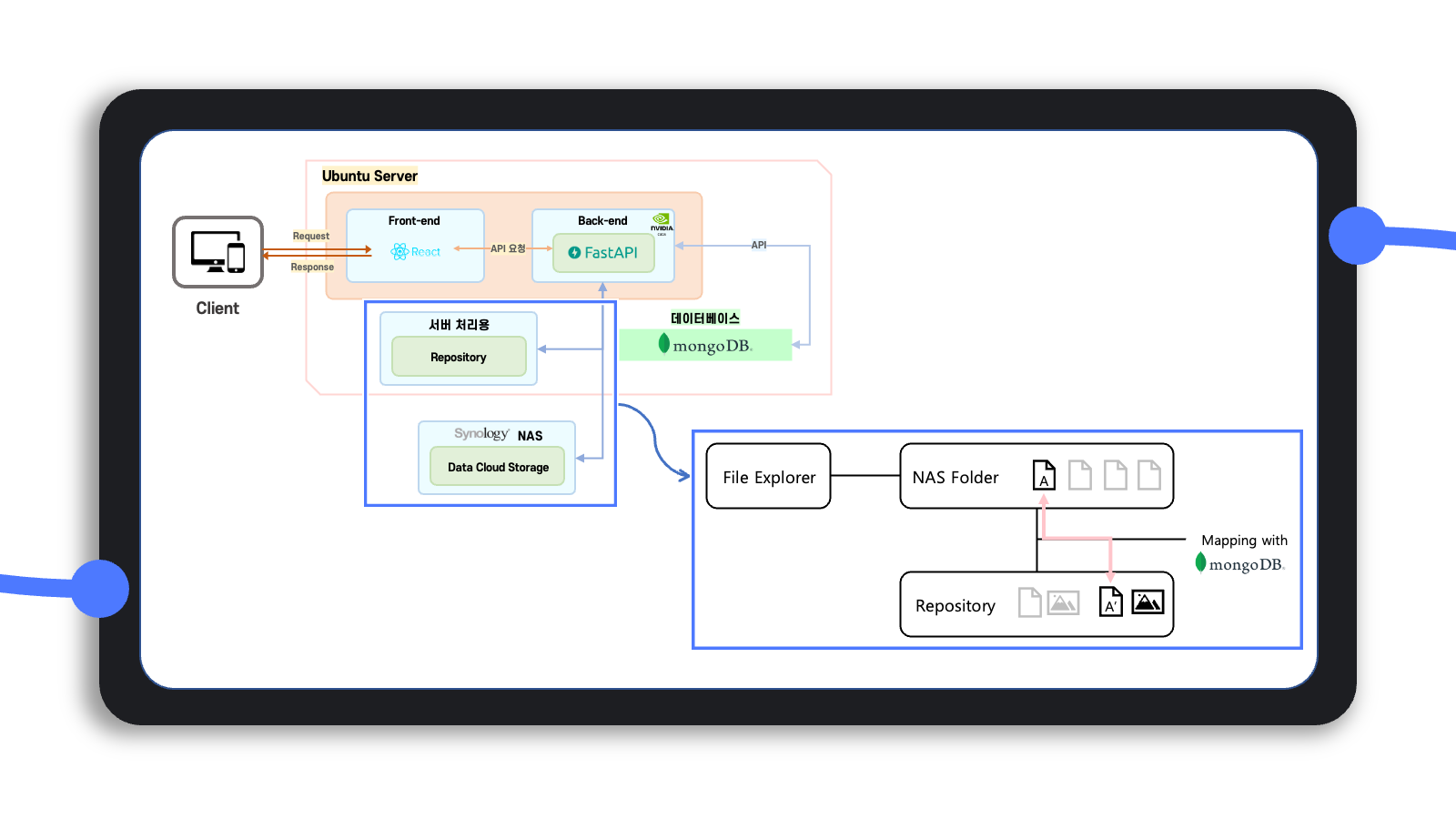
그래서 서버에서 데이터를 처리하면서 진행 결과를 임시로 저장하는 용도의 repository와 사용자의 클라우드 저장소의 데이터를 서로 mapping 시킬 필요가 있었고, 이는 mongoDB를 통해 파일끼리 mapping 할 수 있는 기능을 개발했다.
사용자의 클라우드 저장소에 있는 어떤 한 데이터의 파일 경로를 알면 이를 처리하는 서버에서의 데이터 repository 경로를 알 수 있도록 logic을 구현한 것이다.
Viewer를 통해 대용량 이미지를 원하는 배율로 분석할 수 있다
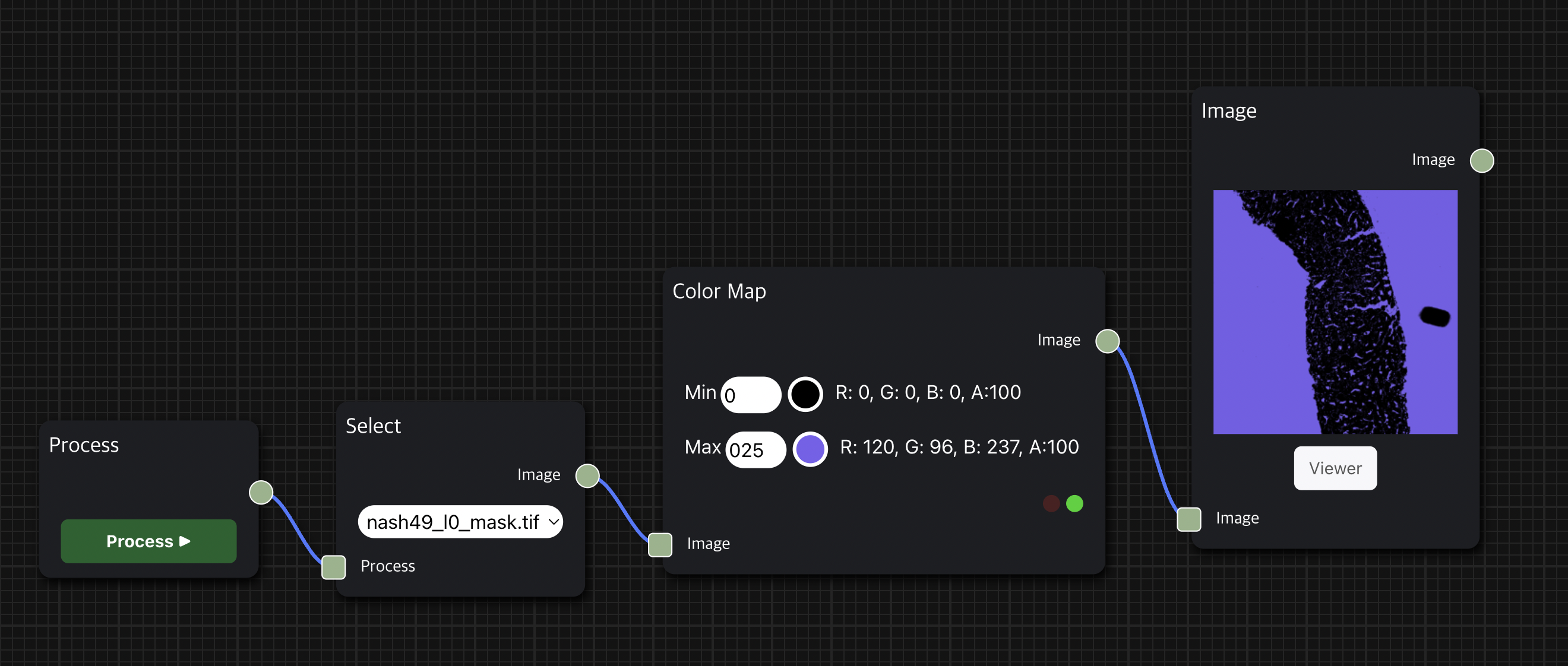
Diagram workspace에서도 분석 결과를 간단한 썸네일로 확인할 수 있지만, 이는 원본 파일이 아니어서 특정 부분을 자세히 분석하는 데는 한계가 있다.
그래서 Viewer 페이지를 통해 사용자에게 면밀한 이미지 분석을 수행할 수 있는 기능을 제공하려고 했다.
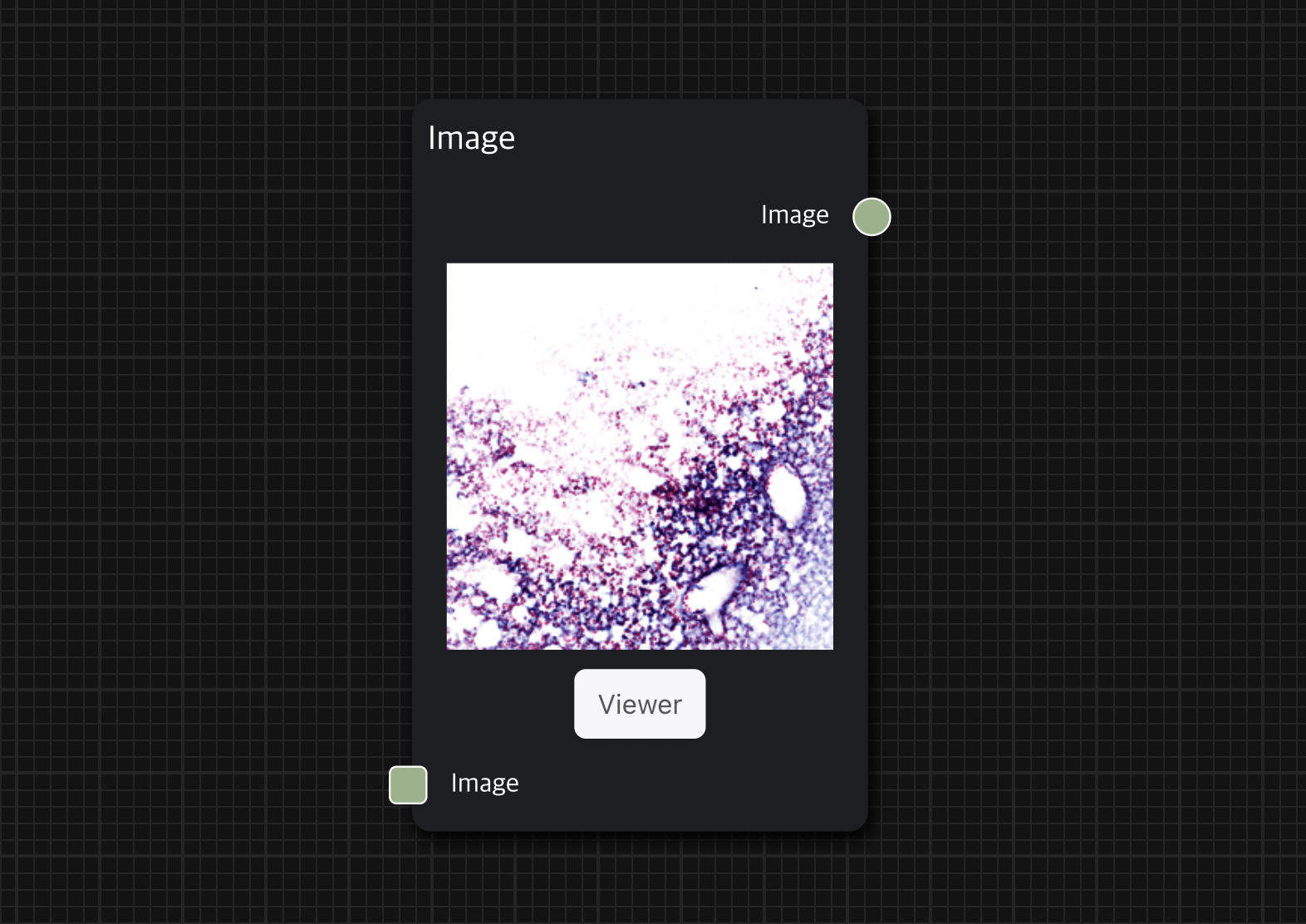
Viewer에서는 이미지를 고해상도로 로딩하여 확대 또는 축소를 통해 이미지를 더 자세히 분석할 수 있다.
Image node의 아래 Viewer 버튼을 클릭하여 해당 이미지의 Viewer로 접속 가능하다.
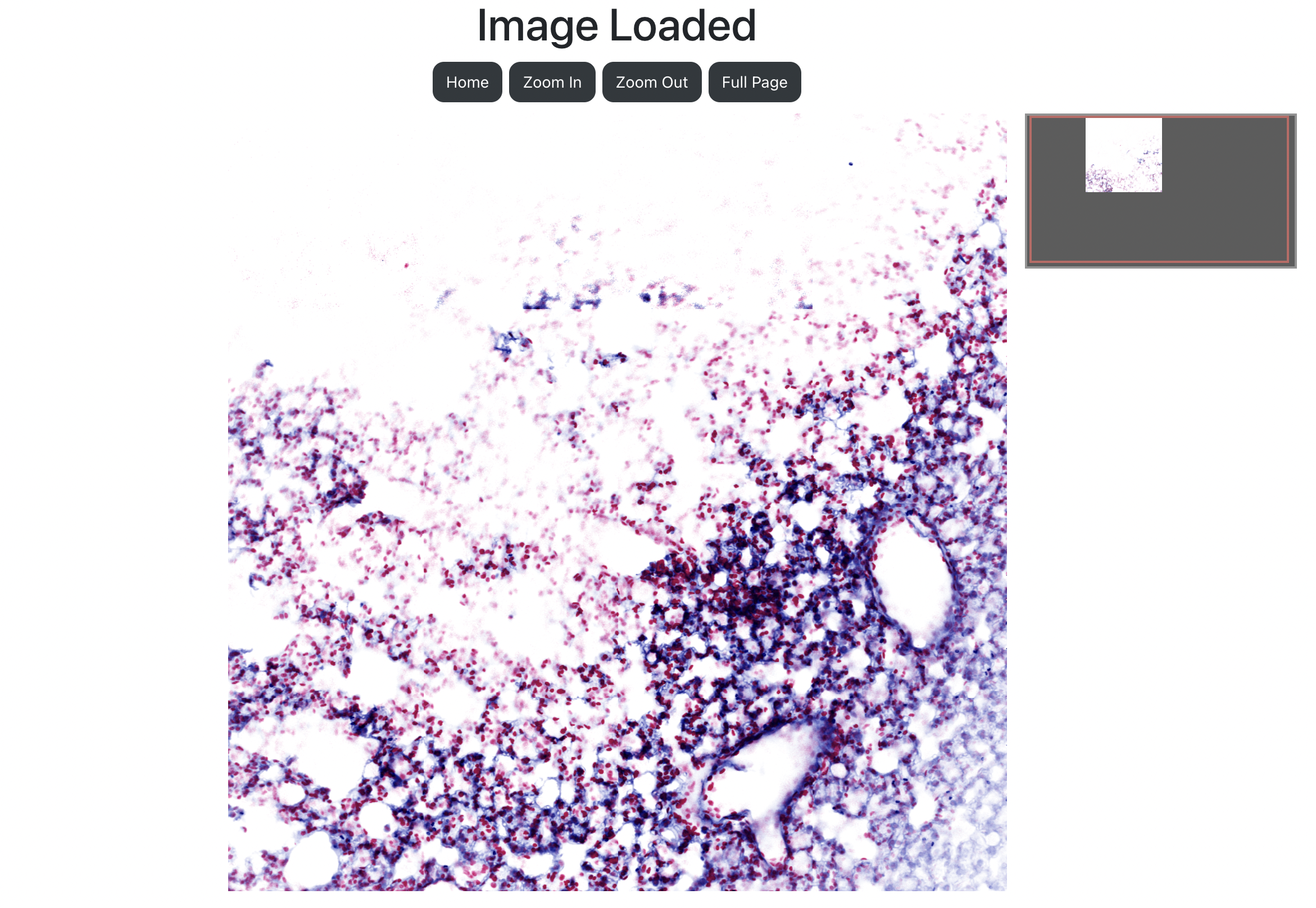
Viewer 페이지는 고해상도의 이미지를 확대하거나 축소할 수 있는 기능을 제공하는 OpenSeadragon 라이브러리를 사용했다.
OpenSeadragon은 워낙 고해상도 2D 이미지를 분석하는 데 있어서 표준인 라이브러리여서 다른 라이브러리를 고민할 필요가 없었다.
OpenSeadragon
OpenSeadragon Viewer With Default Settings Plugins can be used to enhance OpenSeadragon. The following plugins are currently available: OpenSeadragonizer enables viewing any image on a webpage with OpenSeadragon. For installation instructions, see the Gett
openseadragon.github.io
OpenSeadragon을 사용하기 위해서 백엔드에서 OpenSeadragon이 해석할 수 있는 자료구조를 정의해야 하는데, 이는 아래에서 좀 더 자세히 소개하고자 한다.

마우스 포인트가 가리키는 영역을 확대 또는 축소하고 싶으면 원하는 영역으로 마우스를 옮겨서 스크롤하거나 트랙패드로 Zoom In 또는 Zoom Out할 수 있으며, 오른쪽 상단에 미니 맵(Mini Map)을 추가하여 사용자가 보고 있는 영역이 전체 이미지에 어느 부분인지를 확인할 수 있다.
프로그램 개발 과정에서의 노력과 시도
대용량의 이미지 데이터를 처리하는 방법
일반적인 이미지 데이터를 사용한다면 문제가 되지 않았겠지만, 의료 영상 자료는 현미경 또는 고성능 카메라로 세포 조직을 확대하여 자세히 촬영한 경우가 많다.
세포 조직 내에서 어떠한 부분에서 암이 의심되는지 또는 어떤 영역이 병리학적으로 이상 징후를 보이는지를 영상 자료 분석을 통해 알 수 있어야 하므로 coarse한 특징뿐만이 아니라 dense한 특징도 분석이 가능해야 한다.
그러므로 대개 의료 영상 자료는
사실 이로 인해 biomedical 분야에서도 큰 이미지를 처리할 수 있는 효율적인 자료구조를 설계하는 일은 흔한 일이며, 이번 프로젝트에서도 대용량 이미지를 처리할 수 있는 새로운 자료구조의 필요성을 느꼈다.
또한 OpenSeadragon이 해석할 수 있도록 서버에서 처리한 결과를 전송해야 했는데, 이를 충족하려면 결국 OpenSeadragon에 맞게 새로운 자료구조를 정의할 수 밖에 없었다.
DeepZoom Image
먼저 이미지를 patch 단위로 잘라서 관리하는 파일 형식을 탐색했는데, 그중에서 IIIF와 DZI(DeepZoom Image) 두 가지를 후보로 골랐다.
IIIF는 국제 이미지 호환 프레임워크이며, 이미지 서버에 관한 python code가 공개되어 있다.
그러나 이를 응용프로그램에 가져다 쓸 수 있기보다는 이미지를 관리하는 서버로 코드가 작성되어 있어서 활용하기가 불편했다.
DeepZoom Image는 이미지를 효율적으로 처리하기 위해 Microsoft에서 개발했던 Silverlight를 지원하는 기술이며, 입력으로 받은 이미지를 DZI 파일 형식으로 변환하는 python code가 공개되어 있다.
백엔드를 기본적으로 FastAPI로 작성하고 있었고, 기능 개발을 위해 사용한 딥 러닝 모델이 python 코드 기반이어서 백엔드에 바로 응용하여 커스터마이징 할 수 있는 DeepZoom Image가 좀 더 효율적이라고 판단되어 결국 DeepZoom Image를 선택했다.
아래는 python으로 DZI를 변환하는 코드를 살펴볼 수 있는 Github Repository이다.
Python으로 DeepZoom Image를 만드는 Tool
GitHub - openzoom/deepzoom.py: Python Deep Zoom Tools
Python Deep Zoom Tools. Contribute to openzoom/deepzoom.py development by creating an account on GitHub.
github.com
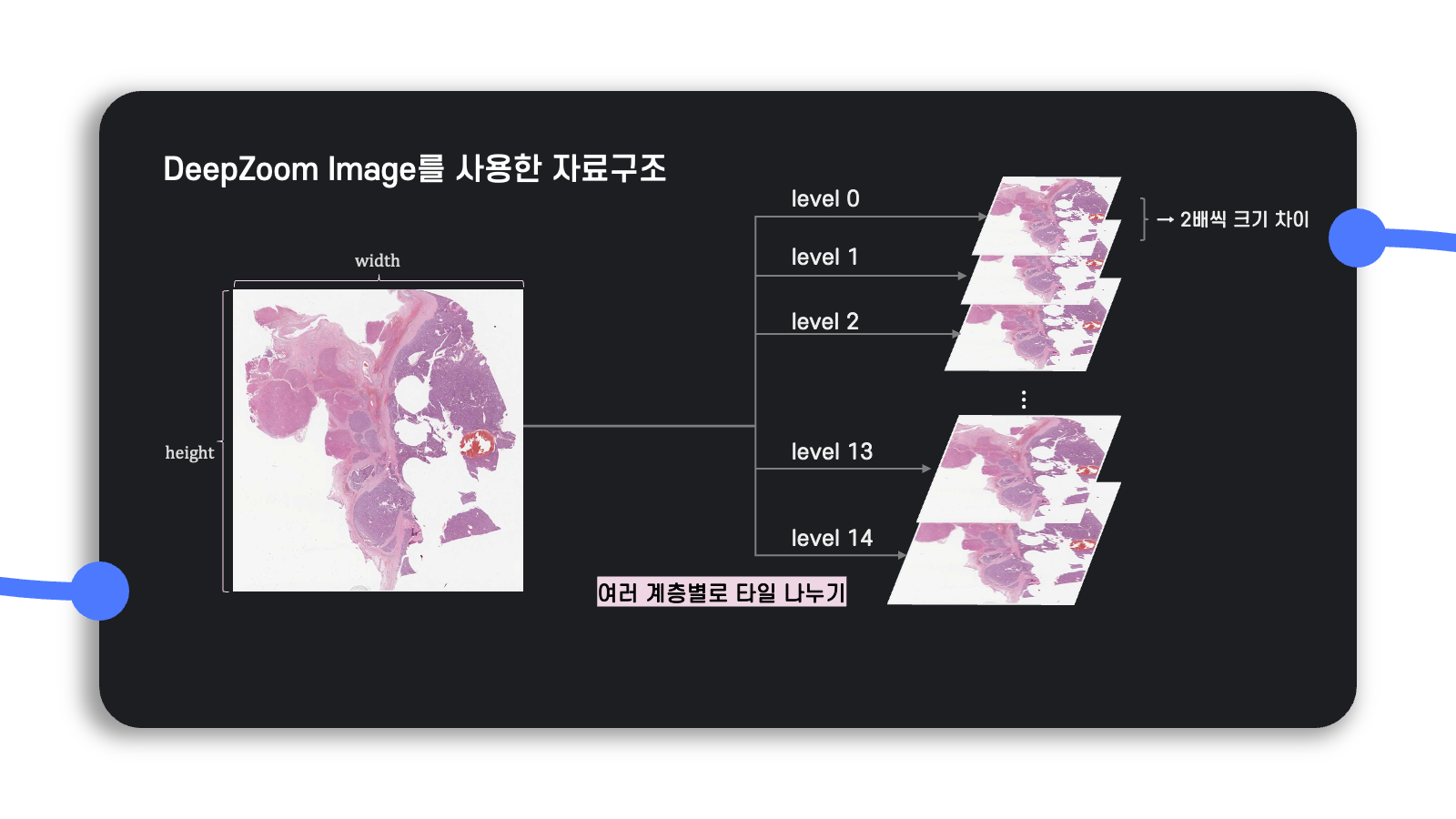
DeepZoom Image는 원본 이미지를 두 배 확대한 사진부터 시작해서
Level 값이 작아지면 작아질수록 원본 이미지에
그리고
예를 들어,
그러면 처음에
그러므로
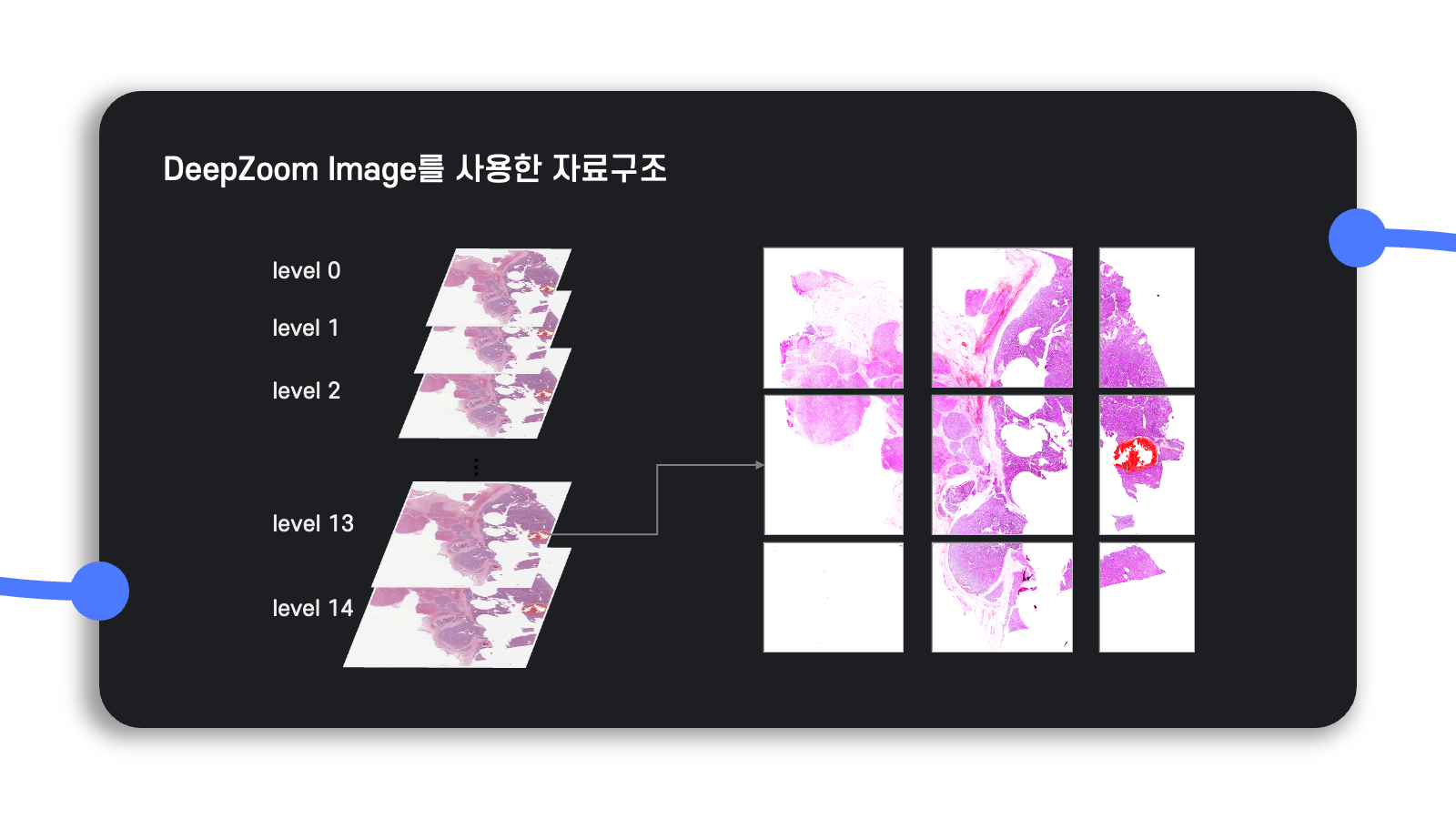
이렇게 생성한 DZI level에서 각 level마다 일정한 크기의 tile(patch)로 자른다.
Tile의 크기는 어떠한 레벨에서든지 동일하므로 가장 해상도가 높은 level 14에서 더 많은 tile이 생성될 것이다.
단, 이미지의 크기가 항상 tile 크기의 배수로 나누어 떨어지는 것은 아니므로 가로 또는 세로의 남는 부분이 존재하면 남는 크기만큼 하나의 tile로 생성한다.
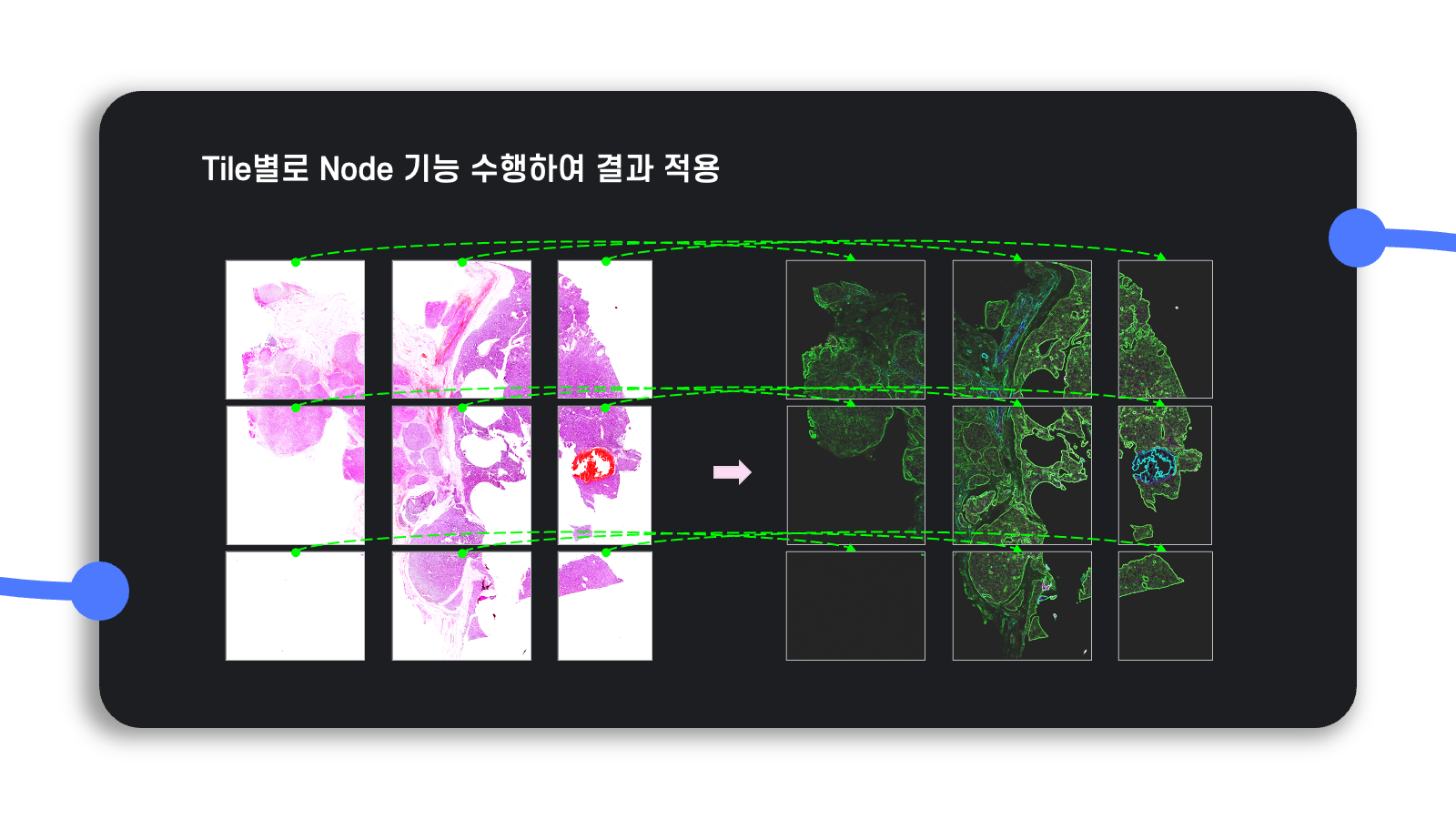
이처럼 tile로 나눠서 이미지를 관리하면 대용량의 이미지를 메모리에 한꺼번에 올릴 필요 없이 tile별로 원하는 workflow를 수행할 수 있으므로, 시간은 좀 더 걸릴지언정 효율적인 메모리 관리를 보장할 수 있게 된다.
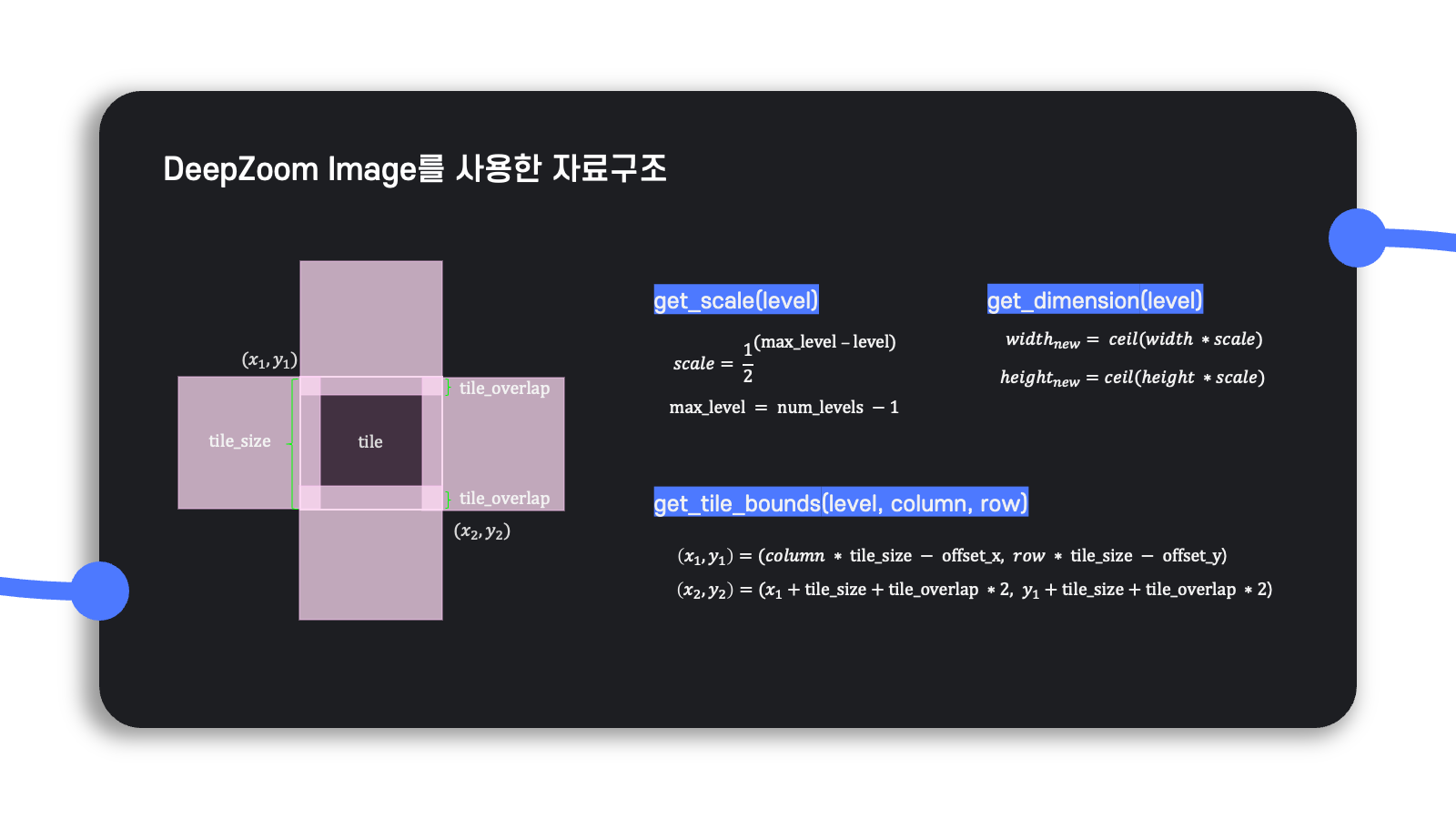
앞서 소개했던 DeepZoom Image로 변환하는 python code에는 변환 logic을 위한 여러 method가 정의되어 있는데, 이를 그림으로 도식화해보면 위와 같다.
놓치지 말아야 할 점은 overlap인데, 이는 tile을 overlap 없이 처리하는 경우도 존재할 수 있지만 overlap이 존재하지 않으면 Gaussian blurring 등 일부 이미지 처리 기능을 수행할 때 전체 이미지에서는 가운데에 해당되지만 tile에서는 가장자리 부분에 해당되어 전체 이미지의 의도대로 tile이 처리되지 않을 수 있다.
그래서 overlap을 0이 아닌 값으로 설정하면 두 tile의 겹치는 영역의 평균을 구하거나 최대•최소로 픽셀 값을 바꾸는 방식을 통해 tile별로 처리한 결과가 전체 이미지를 한 번에 처리했을 때의 결과와 최대한 유사해지도록 할 수 있다.

그런데 모든 level에 관해 tile별로 처리한 결과만을 반환하면 이는 의도하지 않은 결과를 가져올 수 있다.
예를 들어, 가장 해상도가 높은 이미지에서
이를 위해 가장 해상도가 높은 level의 tile들은 모두 처리하고, 상위 level의 tile을 만들 때는 이전 하위 level의 같은 위치에 대응되는 최대 4개의 tile을 병합한다.
실행 시간을 줄이고자 한 시도

Tile별로 데이터를 관리하면 고해상도의 대용량 이미지를 처리하는 데 생성되는 tile이 많아지고 각 tile별로 따로 기능을 수행하다보니 전체 이미지를 처리하는 데 연산 시간이 오래 걸린다.
이를 해결하고자 엔지니어링 관점에서 실행 시간을 줄이고 프론트엔드 측면에서 사용자의 경험을 해치지 않기 위해 다음과 같은 방법을 시도했다.
Thumbnail 출력을 위한 flow와 고해상도 작업을 위한 flow 분리
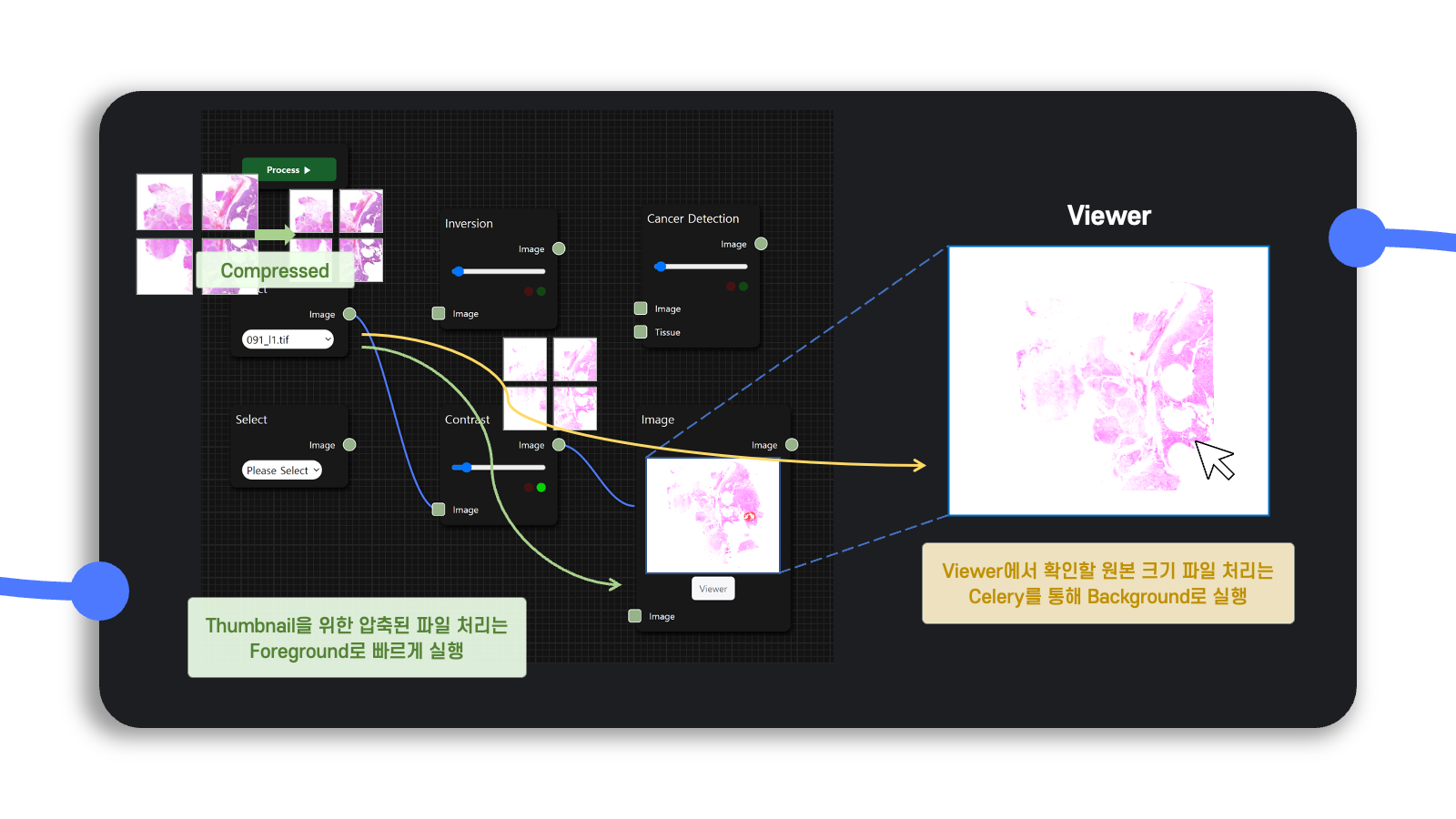
Diagram workspace에서는 현재 사용자가 구성한 flow를 실행할 때 나오는 결과를 간단히 보여줄 필요가 있으며, 이는 빠르게 간단히 보여주기만 하면 된다.
그러나 Viewer를 통해서 분석하는 고해상도 처리 결과는 위에서 설명한 tile별로 관리하는 자료구조로 인해 상대적으로 시간이 더 많이 소요된다.
그래서 thumbnail을 보여주는 용도의 flow는 foreground로 빠르게 처리하고, 고해상도 이미지를 처리하는 flow는 사용자가 요청한 응답을 기다리지 않도록 background로 실행할 수 있도록 작업했다.
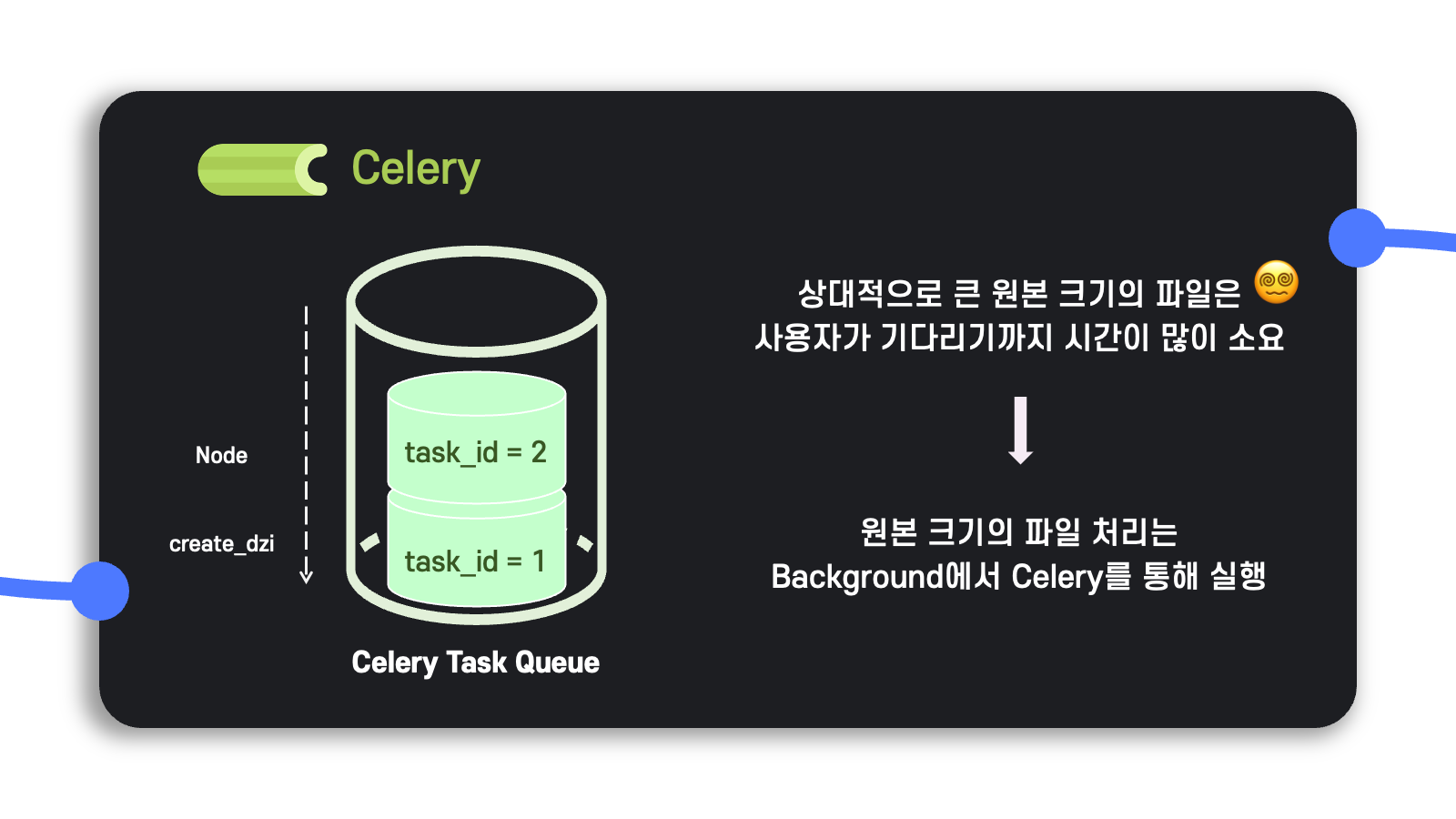
Background의 작업을 사용자의 workflow 순서에 맞게 순차적으로 실행할 수 있도록 task queue를 만드는 것이 필요했는데, 이는 celery라는 라이브러리를 이용했다.
간단히 소개하자면 celery는 broker와 worker로 구성되어 있으며, broker가 서버로부터 어떠한 요청을 받으면 이를 처리할 수 있는 worker에 적재적소로 보내줘서 worker가 적절히 요청을 처리할 수 있도록 한다.
Node별로 진행 상황을 알 수 있는 Signal 추가

Background로 flow를 분리한 것은 좋지만, 아무리 background 작업이라고 해도 사용자가 아무런 힌트 없이 작업 결과를 기다리게 하는 것은 피로감을 일으킬 것 같다는 생각이 들었다.
그래서 각 node마다 background 작업이 진행 중인지 아닌지를 파악할 수 있는 신호를 추가하여 사용자가 무작정 작업을 기다리지 않도록 했다.
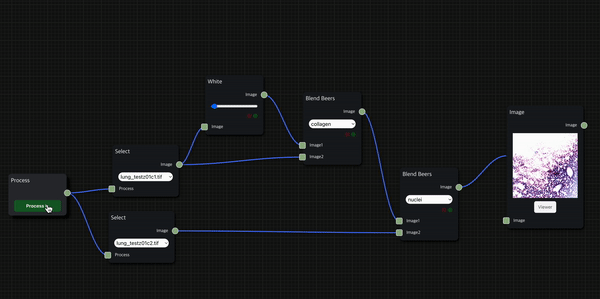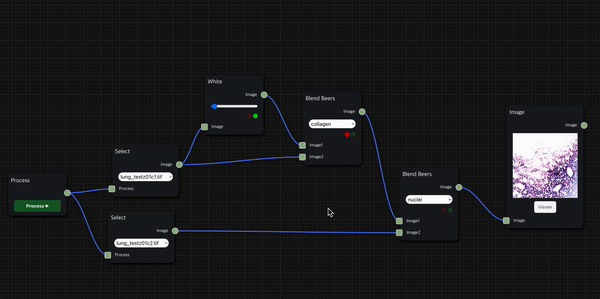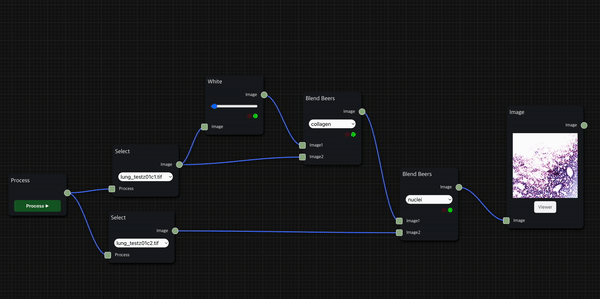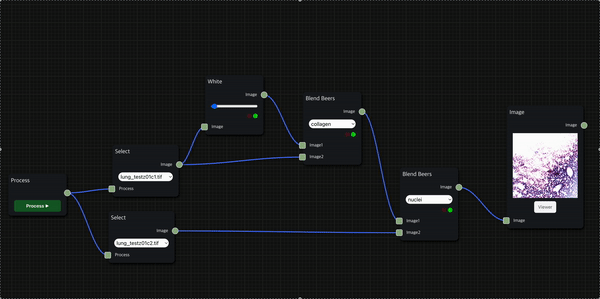
실제로 H&E staining 시나리오를 작동한 모습인데, 상대적으로 시간이 적게 걸리는 foreground에서는 thumbnail을 바로 보여주는 대신 background 작업의 진행 상황은 신호등으로 표시되도록 했다.
Viewer에서의 Streaming
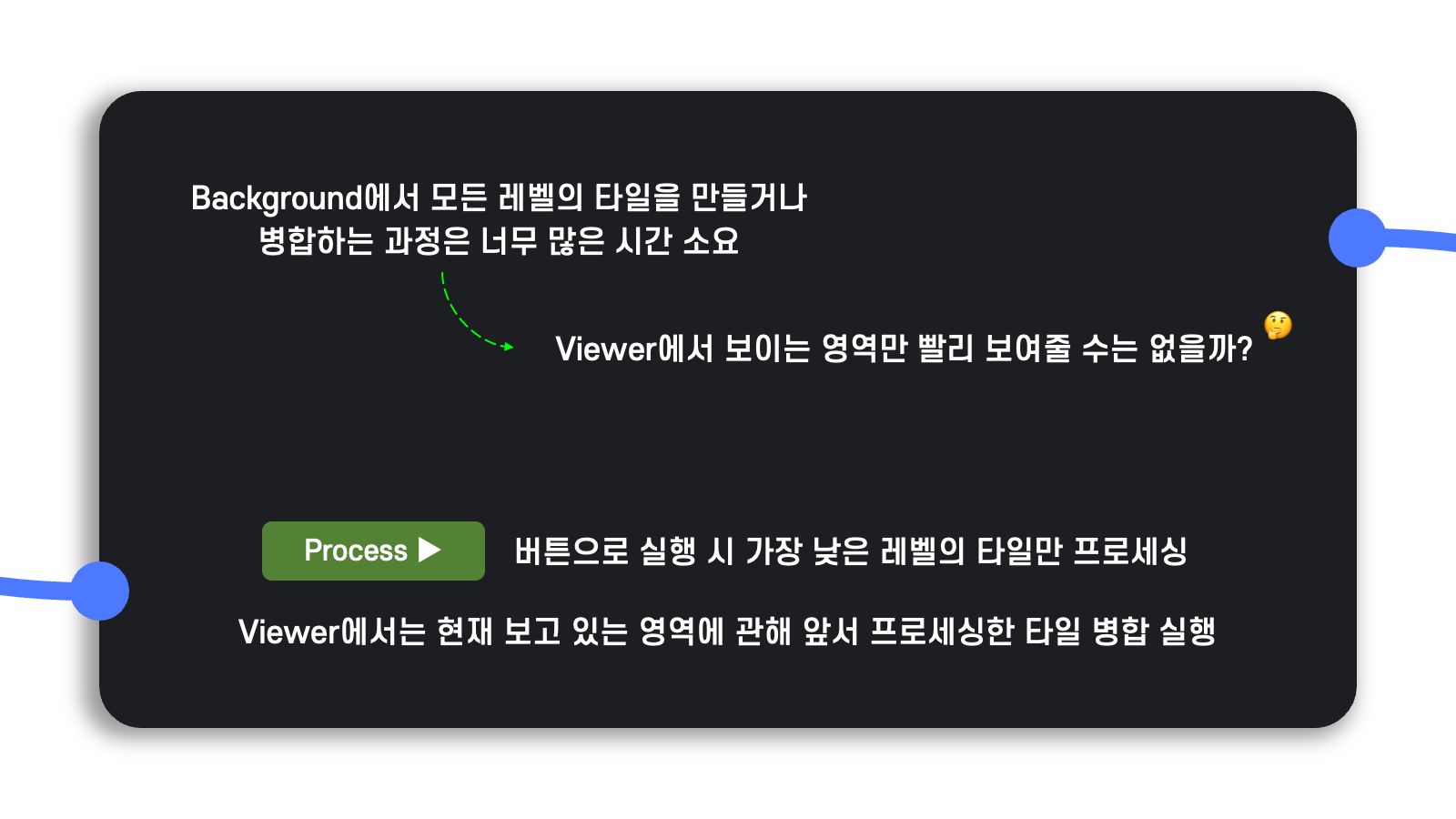
Background 작업을 분리하는 시도가 나쁘진 않았지만, 아무리 background로 수행된다고 하더라도 AI 모델을 실행하는 과정에서 너무나 많은 시간이 소요되었다.
이는 앞서 말한 자료구조의 설명처럼 하나의 고해상도 이미지에 관해 너무나 많은 tile이 생성되어 관리되는 문제 때문인데, 이를 위해 추가로 해결책을 강구했다.
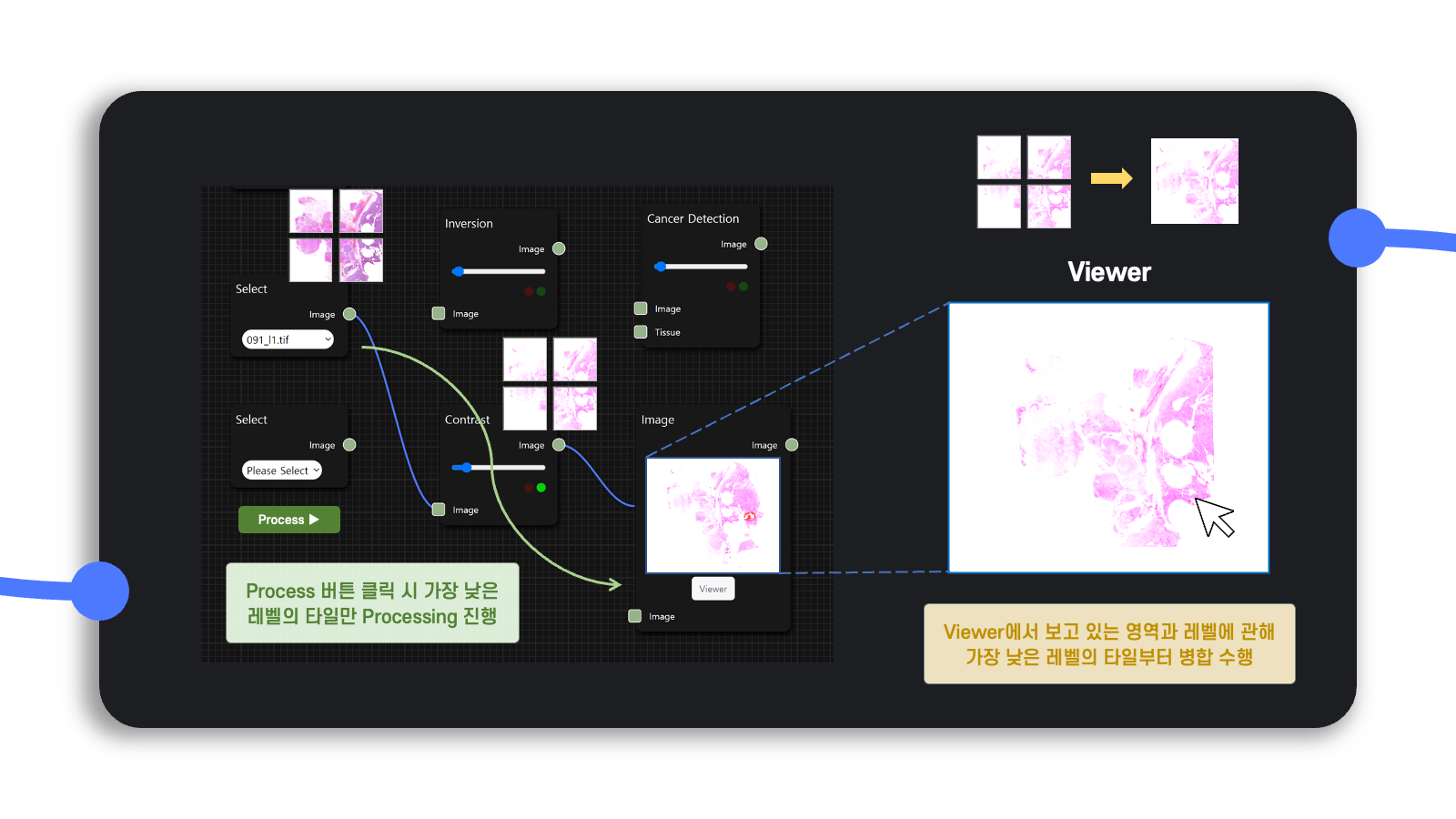
결국 background로 수행되는 작업은 사용자가 Viewer에서 고해상도 이미지를 확대 또는 축소하여 분석하는 데 필요한 것이므로 Viewer에 사용자가 좀 더 이르게 접속할 수 있도록 하여 사용자가 보고 있는 부분만 빠르게 streaming 한다면 소요 시간을 많이 단축할 수 있을 거라고 생각했다.
그래서 diagram의 Process ▶︎ 버튼을 클릭하면 해상도가 높은 가장 낮은 레벨의 타일만 프로세싱을 수행하고, viewer에서는 현재 사용자가 보고 있는 영역에 관해서만 앞서 프로세싱한 타일을 병합하는 작업을 수행하는 방식을 택했다.
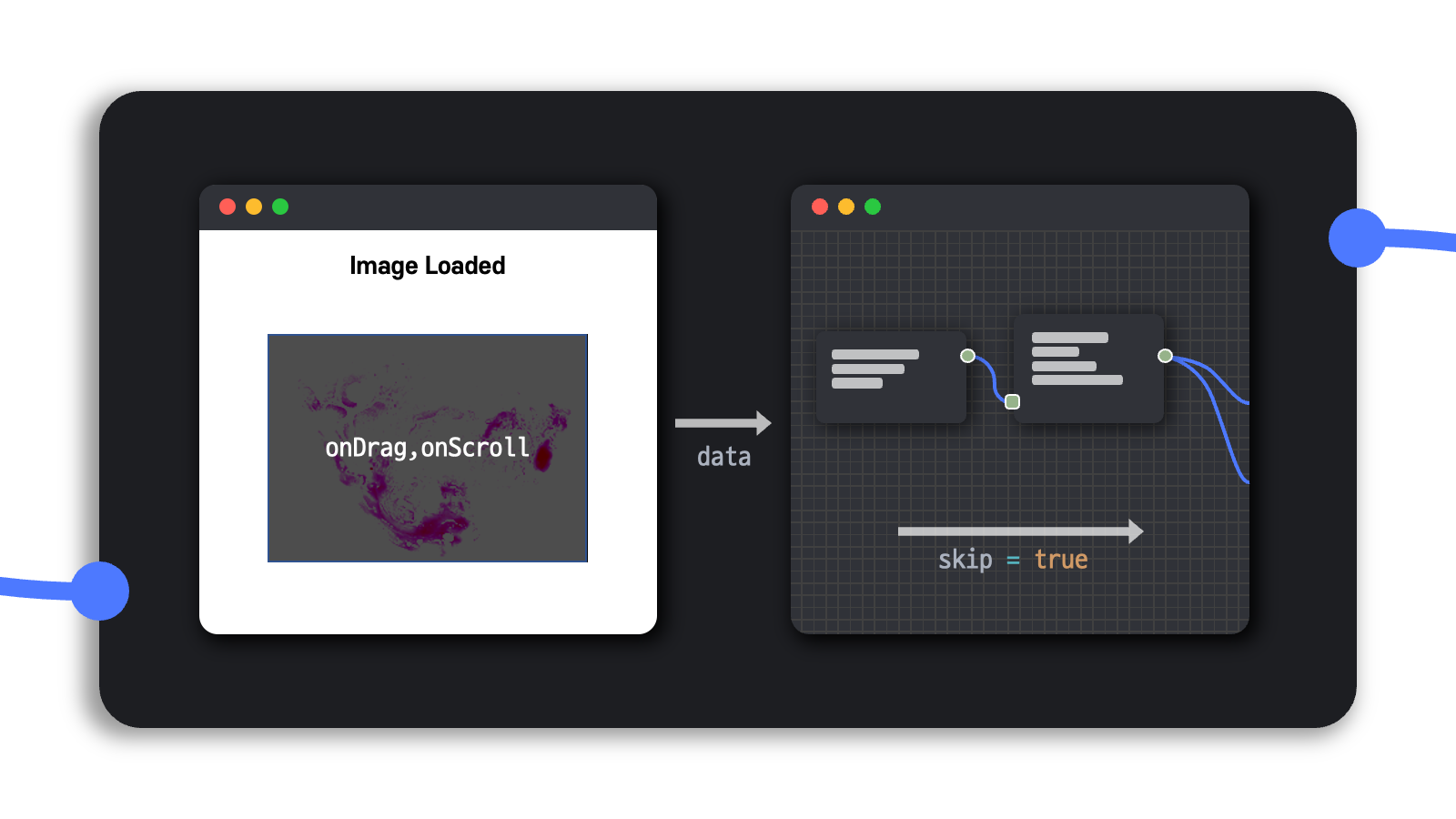
대신에 사용자가 viewer에서 분석 영역을 이동해가면서 이전에 이미 처리한 tile을 다시 보게 될 수도 있으므로 skip이라는 파라미터를 설정하여 이전에 이미 생성한 tile에 관해 연속하여 처리하지 않도록 조치했다.
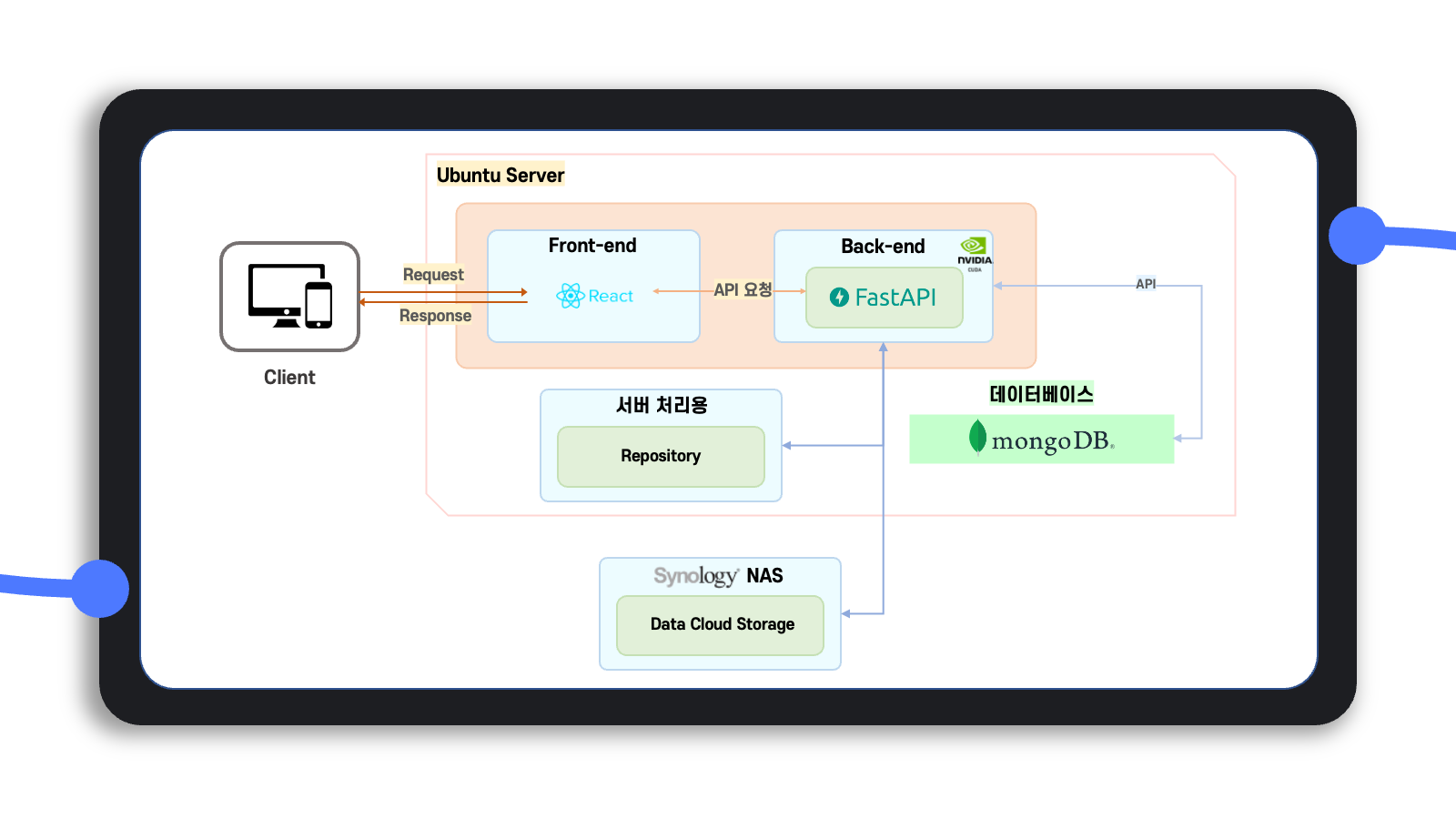
프로그램 Architecture
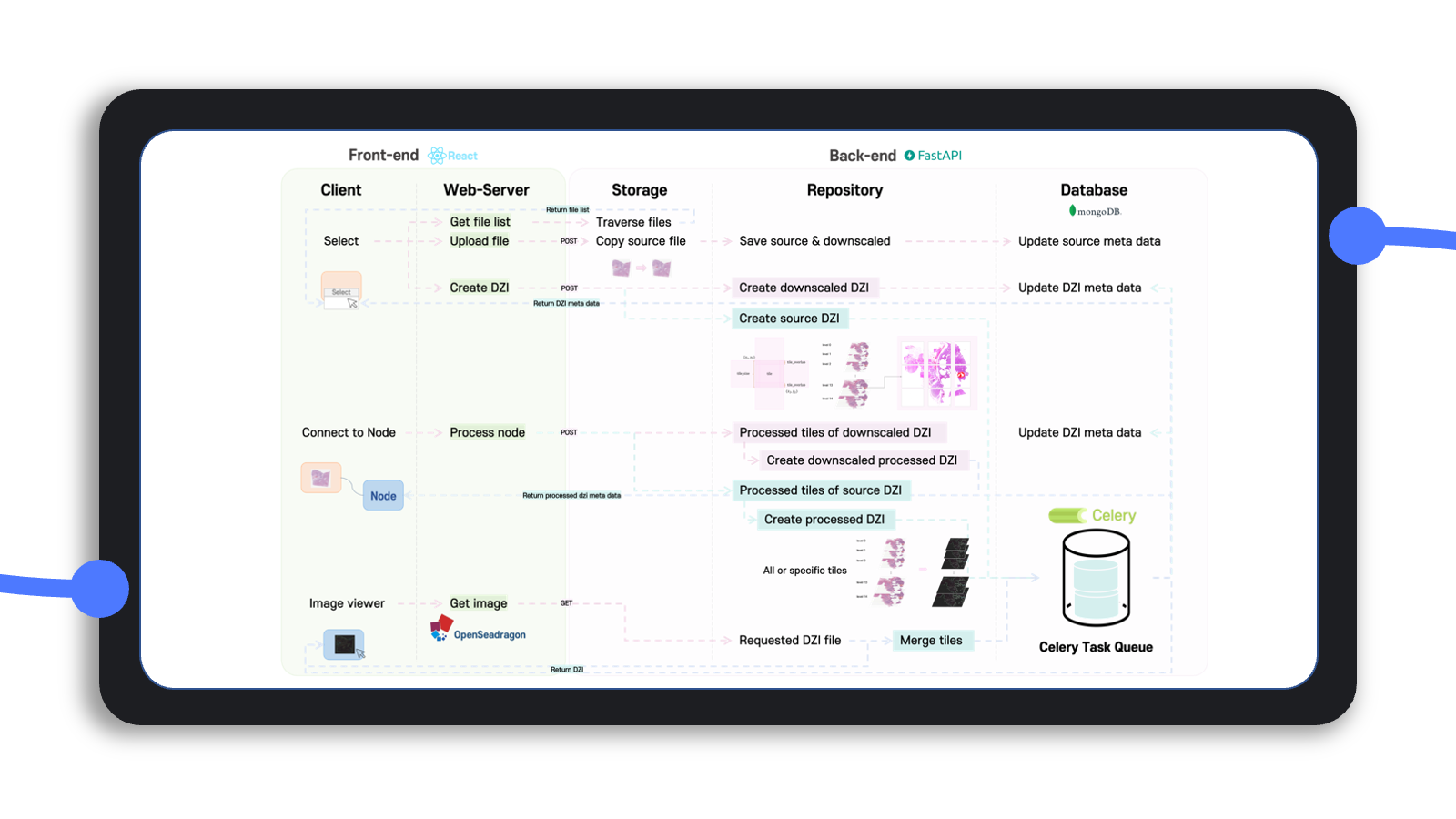
전반적인 architecture와 실행 workflow를 그림으로 정리하면 위와 같이 정리할 수 있다.
요약하면 프론트엔드는 React 기반의 Rete.js를, 백엔드는 FastAPI와 Celery를 사용했으며, 백엔드에서는 사용자의 클라우드 저장소인 storage와 이를 가져와 서버에서 처리하여 임시로 결과를 저장하는 repository, 그리고 storage와 repository를 mapping하는 database를 사용한다.
프로그램 실행 예시
암 영역 진단(Diagnosis of Cancer)
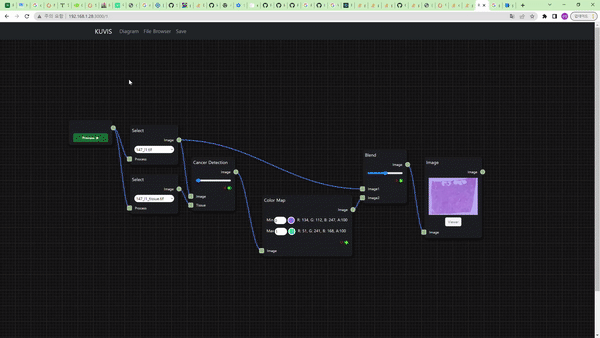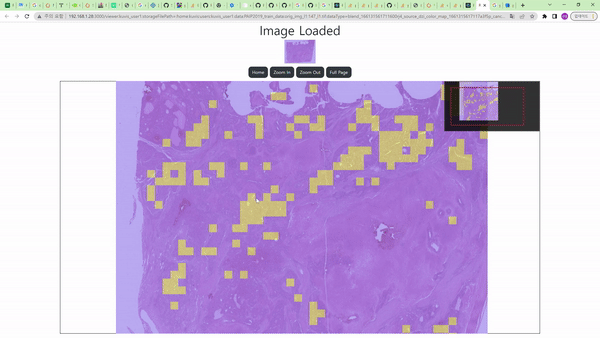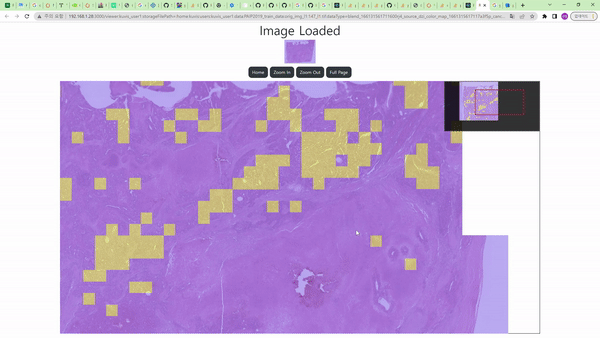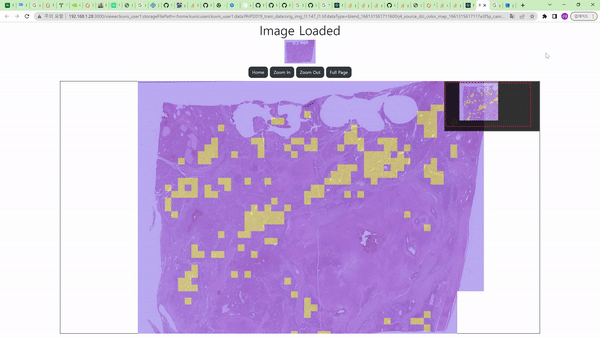
Cancer Detection node를 통해 histopathology의 조직 이미지 데이터에서 암으로 의심되는 영역을 찾는 task를 수행할 수 있다.
256×256 tile size의 patch 단위로 학습된 CNN(Convolutional Neural Network) 기반의 EfficientNet B0 모델을 사용하여 inference를 수행한다.
Image Node와 연결 시 흰색 영역이 암으로 진단된 영역, 검은색 영역이 그렇지 않은 영역으로 보이며, 위의 예시에서는 Color Map node를 거친 결과를 보여주므로 노란색 영역이 암으로 의심된다고 진단한 부분이다.
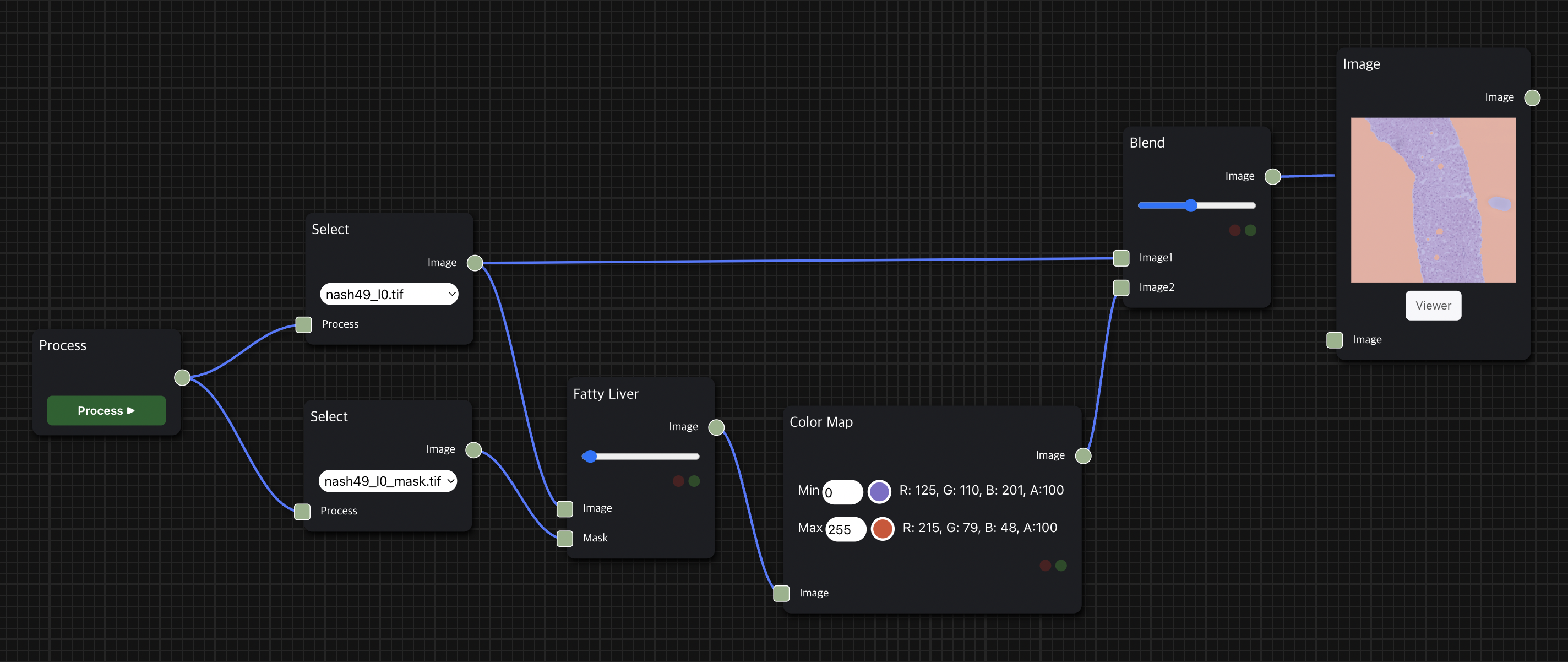
지방간 Segmentation
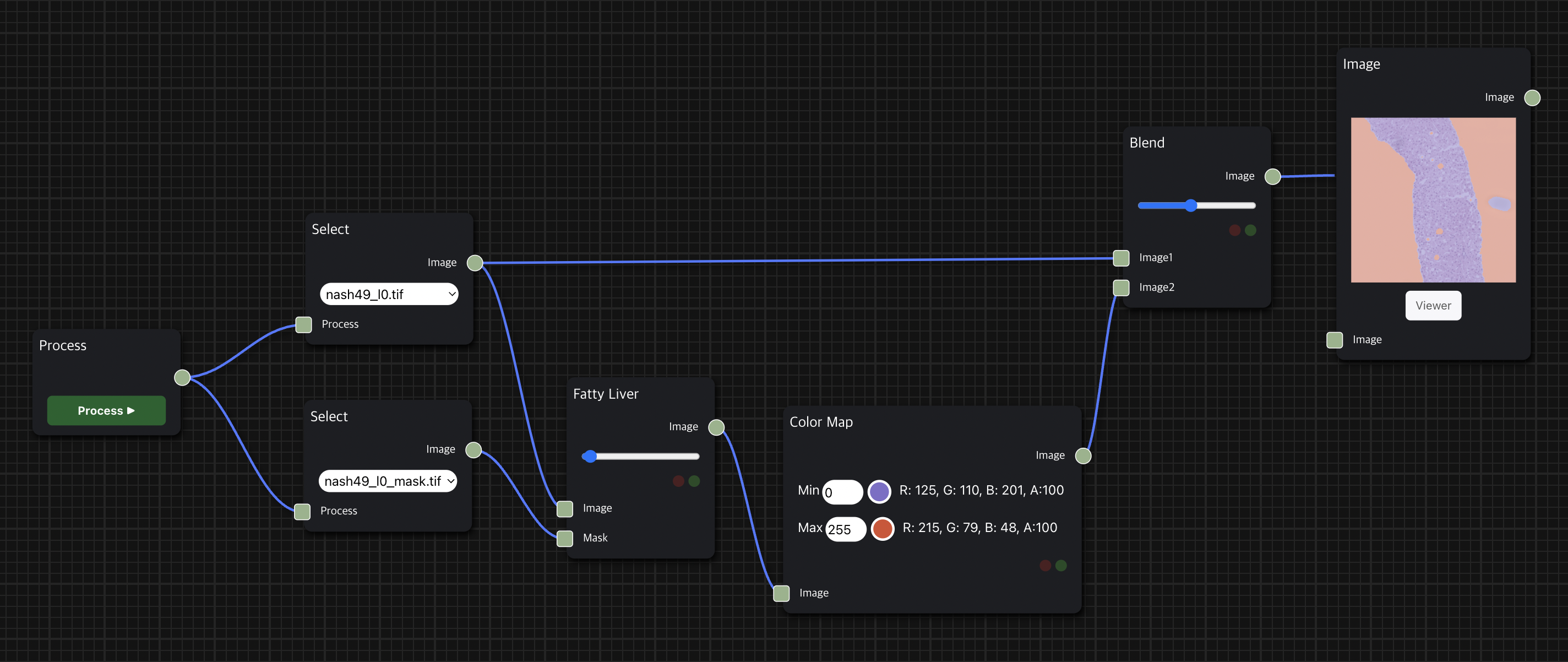
Fatty Liver node를 통해 histopathology의 조직 이미지 데이터에서 지방간으로 의심되는 영역을 찾는 task를 수행한다.
Image Segmentation를 위해 Pixel 단위로 사전 학습되었으며 EfficientNet B4를 encoder로 사용하는 autoencoder 기반의 UNet 모델을 사용하여 inference를 수행한다.
Image Node와 연결 시 세포 조직 내에서 흰색 영역이 지방간으로 의심되는 영역, 검은색 영역은 세포 조직 영역이며, 위의 예시에서는 Color Map node를 거친 결과를 보여주므로 조직에서 비어 있는 것처럼 보이는 주황색 부분이 지방간으로 의심되는 부분이다
.
H&E Staining
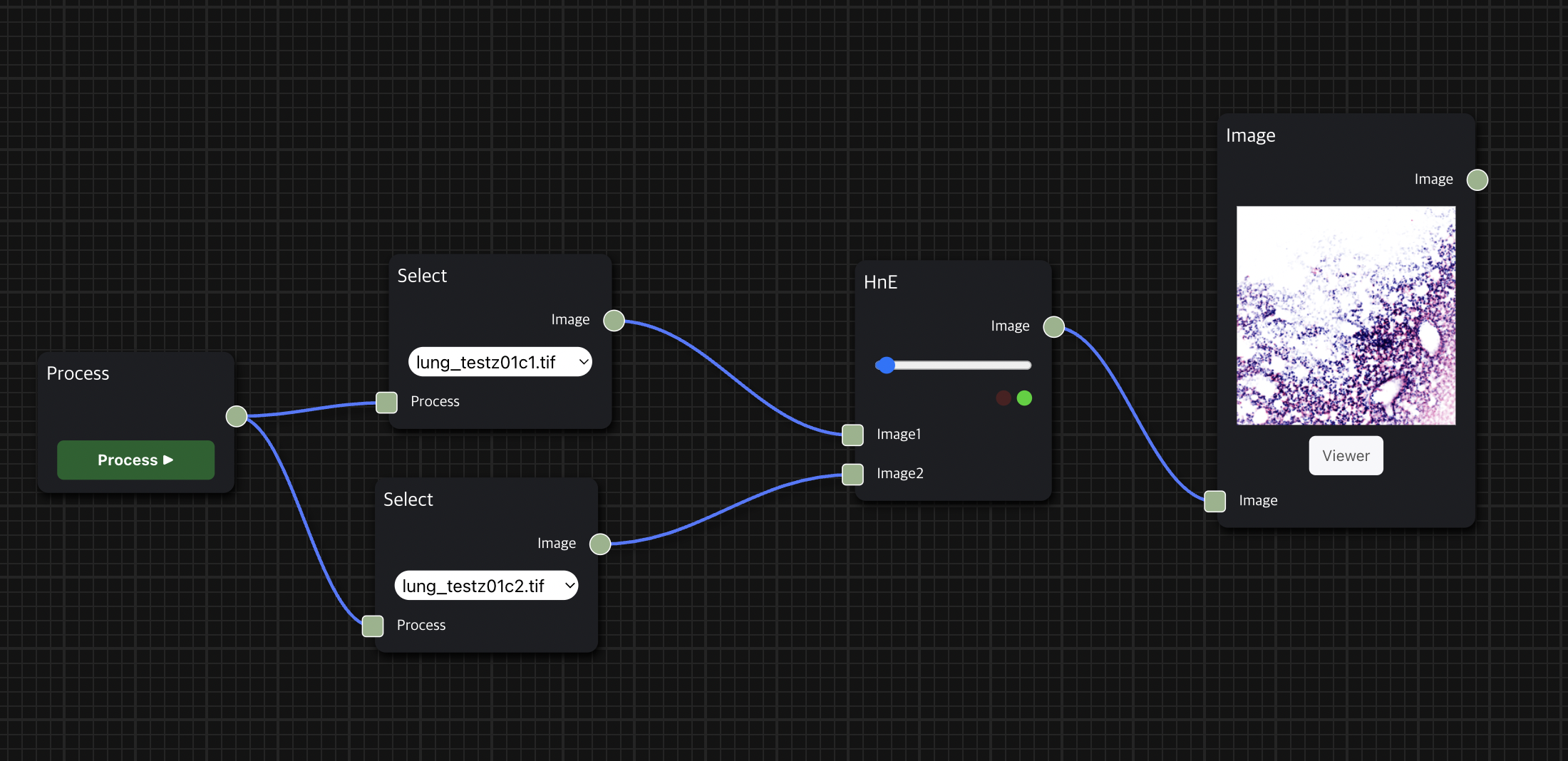
HnE node를 통해 두 개의 histopathology 이미지 데이터를 input으로 사용하여 의학 진단에서 널리 사용되는 H&E Staining을 수행한 결과를 보여줄 수 있다.
첫 번째 input으로는 붉은 색을 띄는 eosin 염색을 적용할 수 있는 세포질 이미지 데이터를 입력으로 받고, 두 번째 input으로는 검푸른 색을 띄는 hematoxylin 염색을 적용할 수 있는 핵(nuclei) 이미지 데이터를 입력으로 받는다.
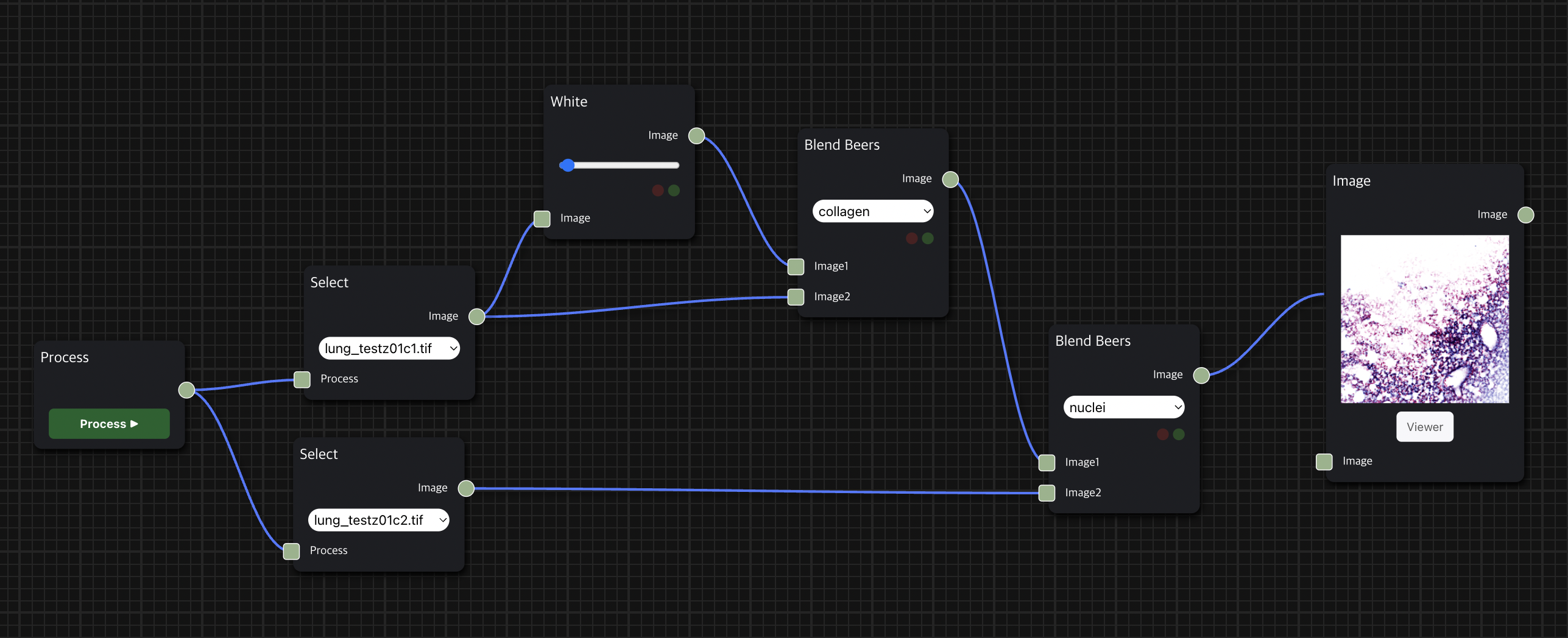
앞서 소개한 H&E 염색 workflow를 위처럼 세분화하여 구성할 수도 있으며, Blend Beers node를 통해 원하는 염색의 종류를 적용하는 필터링을 수행할 수 있다.
발전 가능성
이처럼 이번 프로젝트를 통해 의학 분야에서의 AI 모델링을 웹 기반으로 코드 없이 DAG workflow를 구성하여 사용자의 의도에 맞게 시각화하여 손쉽게 눈으로 확인할 수 있는 어플리케이션을 개발했다.
기본적인 목표는 두 달 동안 달성했고 속도 개선을 위한 여러 시도를 적용했지만, 아직까지도 실행 속도가 빠른 편이 아니라서 아쉽다는 생각이 든다.
Celery를 통해 parallel processing을 구현하고 자료구조로 변환하는 데 불필요한 logic을 제거한다면 속도를 좀 더 향상시킬 수 있지 않을까 하는 생각이 든다.
그리고 이번 프로젝트에서는 pre-trained 모델을 inference하는 기능만 구현했는데, 모델을 직접 선택하여 training하고 evaluation하는 기능까지 추가한다면 더 나은 프로그램으로 발전할 수 있지 않을까 생각한다.
더 나아가 사용자가 직접 원하는 logic의 코드를 작성하여 이를 workflow의 node로 추가할 수 있다면 획기적이지 않을까 하는 얘기가 오고 갔다.
프로젝트 과정에서 참고한 논문
A Survey of Visualization Pipelines

인턴 근무하면서 힘들었지만 재밌고 알찬 방학이 되었던 것 같아서 뿌듯하고 보람차다. 😆
본 프로젝트는 고려대학교 정보대학 HVCL(고성능 비주얼 컴퓨팅 연구실)에서 인턴으로 근무하며 개발한 결과물이며, Vience(바이언스)에 귀속되는 프로그램입니다.
소중한 공감 감사합니다.